Diagnóstico, tratamiento y vacunas

Lluis Montoliu, investigador del Centro Nacional de Biotecnología (CNB-CSIC) ha explicado los últimos avances en la aplicación de la tecnología CRISPR al diagnóstico virológico del SARSCoV2.

Ha comenzado contando el descubrimiento de CRISPR, con una mención obligaba a Francis Mojica, la variabilidad de los distintos sistemas CRISPR y su tremenda versatilidad. La versión 1.0 de CRISPR han sido herramientas genéticas para detectar, cortar y editar secuencias de ADN, pero desde el año 2017 se han ido desarrollando nuevas versiones de CRISPR (versión 2.0) capaces de cortan ARN de forma inespecífica y con nuevas aplicaciones. Bajo distintos acrónimos, cada uno más curioso, se han ido desarrollado nuevos sistemas basados en diferentes tipos de proteínas Cas que se están aplicando para la detección de secuencias virales. Por ejemplo, los sistemas SHERLOCK (CRISPR-Cas13a) que permite ARN con una sensibilidad a nivel atomolar; DETECTR (CRISPR-Cas12a) para cortar ADN de cadena sencilla; CARMEN que combina el sistema SHERLOCK con tecnología microfluídica de nanogotas para ensayos masivos; CONAN (CRISPR-Cas3) capaz de cortar ADN de forma inespecífica. Estos sistemas permiten desarrollar métodos de detección de virus sencillos y rápidos tipo “point of care”.
En comparación con el diagnóstico por RT-PCR, el sistema CRISPR DETECTR, en concreto, es más rápido (menos de 30 minutos) y sencillo, (solo necesitan reactivos, pipetas y un termobloque), aunque son menos sensible y no es cuantitativo. Por ello, DETECTR puede ser un método excelente de cribado poblacional. Aunque todavía no se pueden comercializar, el pasado 8 mayo la FDA norteamericana aprobó su uso de emergencia como sistema de detección. Por otra parte, la capacidad del sistema CRISPR-Cas13d de cortar el ARN viral se está ensayando como posible antiviral. Obviamente, uno de los retos es asegurar la especificidad del sistema y que solo destruya el genoma del coronavirus y no otros ARN celulares.
Víctor J. Cid, Catedrático de Microbiología de la Facultad de Farmacia de la Universidad Complutense de Madrid ha presentado un resumen de la investigación y desarrollo de los tratamientos frente al COVID-19. Aunque de momento no existe ningún tratamiento especifico, a día de hoy hay ya 2749 ensayos clínicos en curso (Referencia). Aunque existen diversas estrategias, muchos de los ensayos se basan en reposicionar fármacos ya empleados para otras funciones, basándose en lo que se va conociendo sobre la biología y replicación del virus y sobre su efecto en nuestras células.
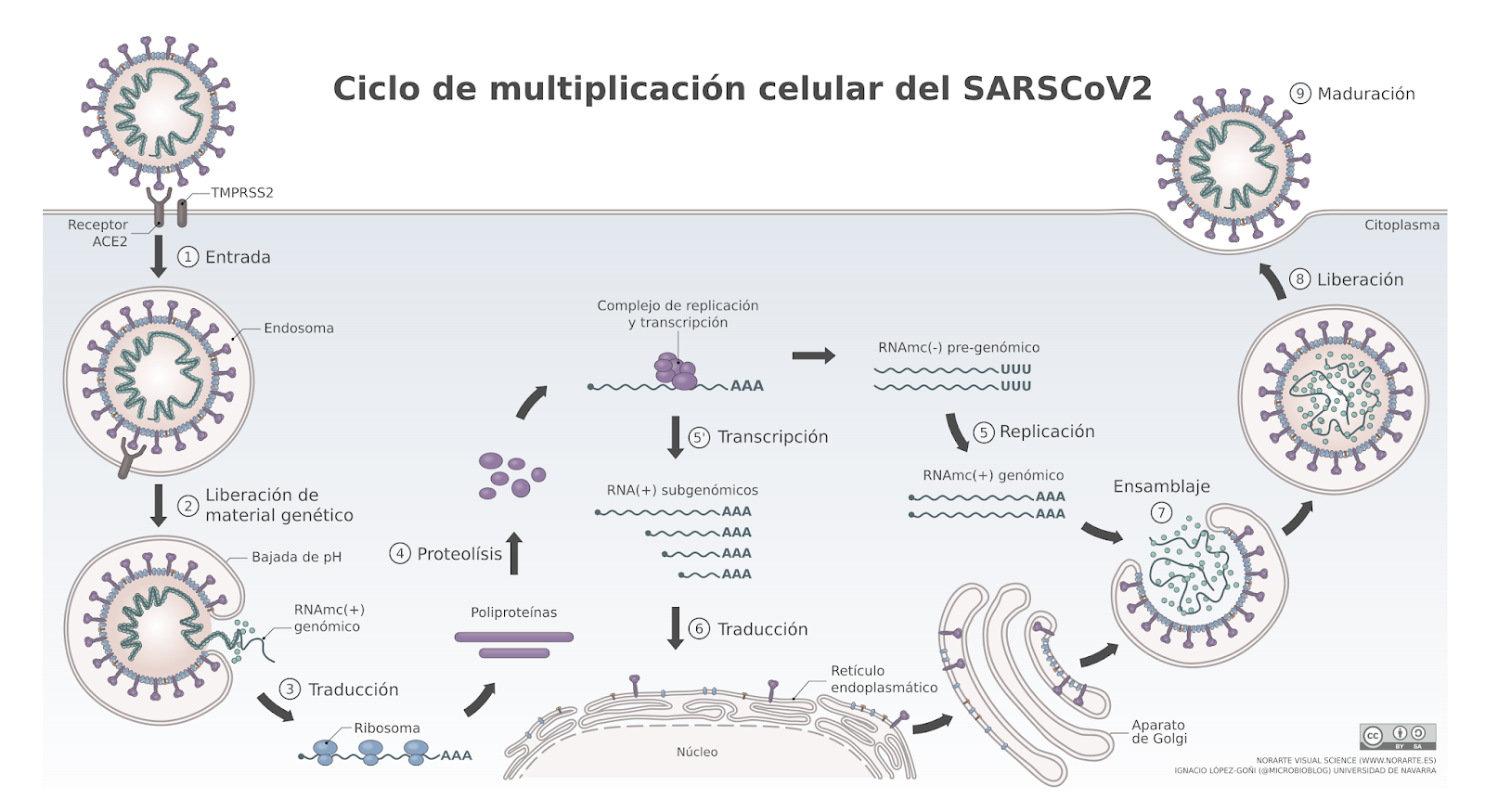
– Inhibir y neutralizar la fase de unión del virus a la célula, a través de la proteína S que se une al receptor celular ACE2 y necesita la acción de determinadas proteasas celulares, como la furina: lectinas que unen azucares para bloquear la espícula S, anticuerpos monoclonales o suero de personas convalecientes (sueroterapia), administración del receptor ACE2, inhibidores de la furina, …
– Fase de fusión de la envoltura del virus y las membranas celulares, que depende de otras proteasas celulares como la TMPRSS2 y de una bajada de pH: inhibidores de las proteasa celulares, compuestos lisosomotrópicos que inhiben el pH, …
– Fase de expresión del ARN, síntesis de poliproteinas y procesamiento posterior: inhibidores de las proteasa virales, …
– Fase de replicación: inhibidores de la RNA polimerasa, helicasa, metiltransferasa viral, …
– Otros fármacos como antiinflamatorios: corticoides, antioxidantes e
distintos inhibidores de la interleuquinas 6, interferones, lactoferrina, ivermectina, etc …
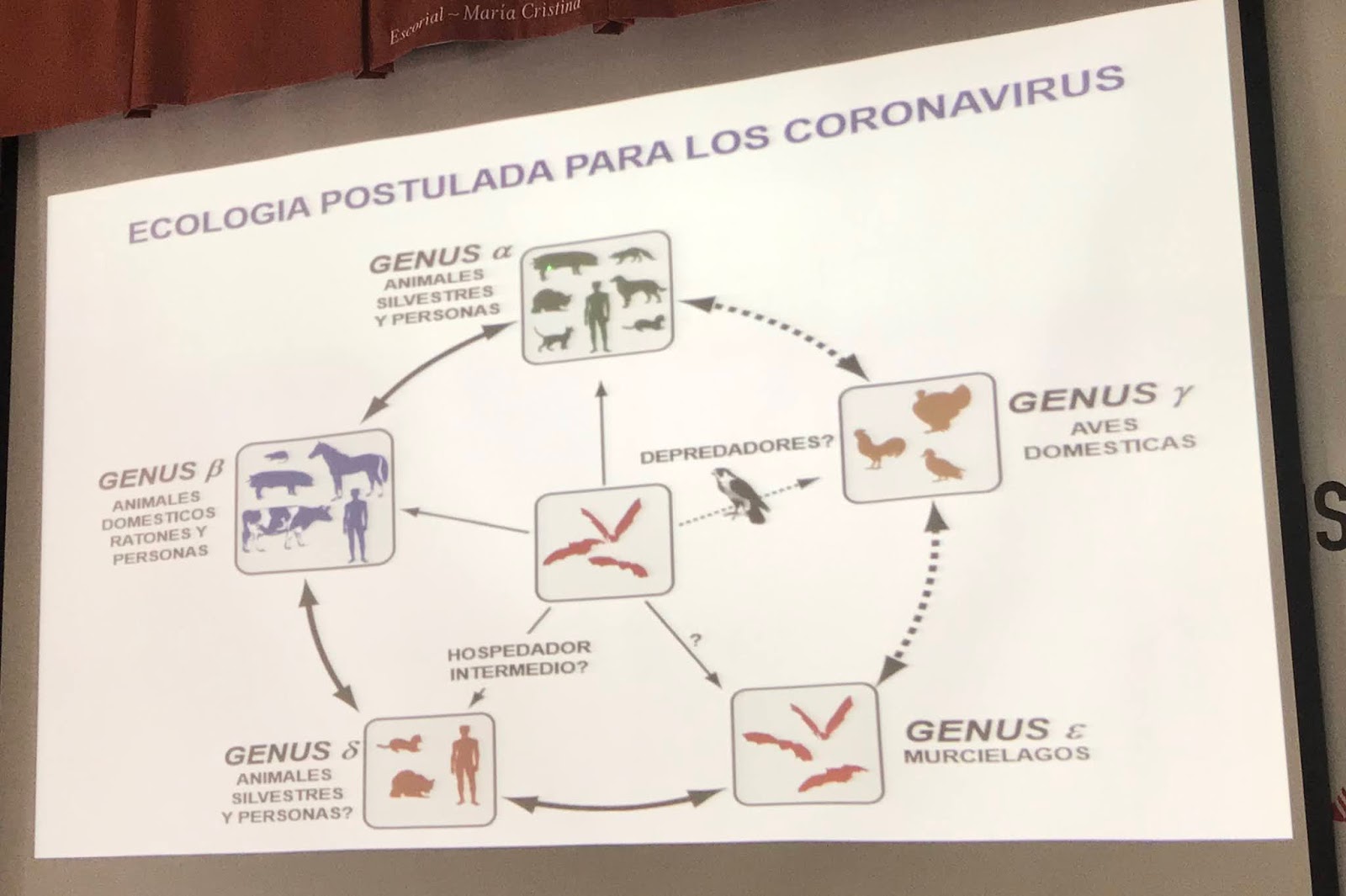
Luis Enjuanes, Profesor de Investigación del CNB-CSIC ha explicado el efecto del cambio climático en la distribución de los virus, con ejemplos concretos de virus transmitidos por mosquitos (arbovirus) como el cambio en la distribución mundial del virus de la lengua azul, el zika o el virus del Nilo Occidental. Respecto a los coronavirus, ha explicado que todos proceden de murciélagos. El paso al ser humano ha ocurrido a través de distintos animales intermediarios, las civetas en el caso del SARCoV1 o los camellos en el caso del MERS (Las civetas de granja se consumen como delicatesen en China, y la leche y orina de camella se beben en Oriente Medio). En el caso del SARSCoV2 sabemos que su origen son también los murciélagos pero todavía no está claro el animal intermediario (se ha sugerido el pangolín y las serpientes, pero no es definitivo).
Probablemente, las cuatro características que hacen que el SARSCoV2 sea un virus muy fácil de diseminarse y de difícil control sean: la presencia del virus en personas asintomáticas que lo pueden transmitir, su enorme capacidad de infectar distintos tipos celulares y causar distintas patologías (en pulmón, intestino, riñón, cerebro, corazón, vasos sanguíneos, páncreas, …), su capacidad de inducir una respuesta inmune limitada, y la reemergencia en un 14% de los infectados en pacientes “recuperados”.
Respecto a la vacuna que se está desarrollando en su laboratorio, han caracterizado los genes de virulencia, esenciales para la patogenicidad del virus. Han reconstruido de forma artificial el genoma del virus, sintetizando fragmentos del genoma y ensamblándolos para generar un cromosoma artificial. Esto les permite obtener una colección de mutantes en los que le faltan desde uno, hasta cinco genes de virulencia (genes 3, 4a, 4b, 5, E). En modelos con SARSCoV1 y MERS, demostraron que estos mutantes no eran virulentos, el virus no se propagaba en las células pero el ARN era capaz de replicarse. No son, por tanto, auténticos virus sino fragmentos de ARN replicantes, replicones. Estos replicones eran capaces de proteger al 100% de los ratones infectados con el virus, actuando como vacunas. Están trabajando en dos prototipos: un basado en nanopartículas como vehículo del ARN replicon y otro en VLPs.

Juan García Arriaza, Investigador Contratado del CNB-CSIC que trabaja en el laboratorio de Mariano Esteban, ha explicado el desarrollo de una vacuna preventiva frente a COVID-19 basada en poxvirus recombinantes. Utilizan como vector el virus vaccinia Ankara modificado (MVA). Se trata de un vector basado en el virus vaccinia que, tras más de 500 pases en el laboratorio, ha ido perdiendo los genes de virulencia, está muy atenuado y no es capaz de replicarse en células humanas, es por tanto muy seguro. Además, tiene otras ventajas: flexibilidad, permite incluirle otros genes; es muy inmunogénico; de fácil de administración y bajo coste de producción. Es un vector ideal, que ya se ha utilizado como vacuna contra la malaria, la tuberculosis, cáncer y otras infecciones virales. En el caso de SARSCoV2, están ensayando dos prototipos de vacunas MVA que expresan la proteína S del coronavirus. En modelo de ratón, las vacunas producen una potente y duradera respuesta humoral (de anticuerpos neutralizantes) y celular (respuesta de alta calidad con distintos tipos de citoquinas). Ahora está pendiente repetir en otros modelos animales (hámster y primates no humanos), y comenzar las fases clínicas (plan previsto: fase I en diciembre 2020, II en marzo-abril 2021 y III en verano 2021).

Adolfo García-Sastre, Co-director del Global Health & Emerging Pathogens Institute y del Icahn School of Medicine at Mount Sinai en Nueva York, ha explicado su estrategia para buscar antivirales frente al SARSCoV2, en colaboración con otros equipos. Su trabajo consiste en buscar posibles inhibidores de las interacciones entre el virus y las proteínas humanas. Para ello se ha basado en estudios de las interacciones entre las proteínas del virus y las proteínas humanas (el interactoma) y en los cambios de fosforilación de las proteínas humanas inducidos por el virus (el fosfoproteoma). Han seleccionado y probado más de 400 compuestos distintos, y han encontrado compuestos sin efecto, con efecto antiviral, en incluso con efecto proviral, in vitro en cultivo celular. El siguiente paso son los estudios en modelos animales y la terapias de combinación. Un compuesto que destaca por su actividad antiviral in vitro es la Aplidina de PharmaMar, que ya ha comenzado los ensayos clínicos.

La mesa redonda se centrado sobre vacunas y se ha unido María Jesús Lamas, Directora de la Agencia Española de Medicamentos y Productos Sanitarios. Algunos comentarios que se han hecho:
Nos interesan vacunas cuanto antes, pero seguras, según cuatro principios básicos: i) no hacer el mal, que lo que se haga no cause otros efectos, ii) hacer el bien, que protejan, iii) información y consentimiento de las personas, iv) que estén a disposición de todo el mundo que lo necesite.
Las prisas no pueden ser excusas para saltarse estos requisitos, se trata de acelerar los pasos pero no saltarse ningún paso, por eso las agencias reguladoras. No se suprimen fases, se solapan. Se están empleando vectores en los que ya había mucha experiencia, regulación y autorizaciones para otros antígenos (adenovirus, poxvirus, …), por eso se va más rápido. Se ha acelerado el proceso de obtención de la vacuna también porque ha habido un aumento de inversión sin precedentes. Habrá varios tipos de vacunas, para distintos usos, distintas edades, se necesitan vacunas que se puedan fabricar y distribuir de forma rápida, que sean seguras y que funcionen, aunque no sean las propuestas más sofisticadas y mejores. Y sin perder la confianza de la población para no favorecer los movimientos antivacunas, por eso es importante la transparencia. Solo se van autorizar vacunas por criterios científicos, se va a ser tan rigurosos como si el desarrollo hubiera llevado 10 años. Se es más exigente en la autorización de vacunas que de cualquier medicamento. Otros medicamentos se administran a personas enfermas, las vacunas, en principio, se administran a personas sanas. Se aprueban según la relación beneficio-riesgo: no hay riesgo cero. Después de su aprobación se sigue con una fase IV de farmacovigilancia, cuando se emplean en cientos de miles de personas muy diferentes. En España tenemos el conocimiento, pero falta cooperación y capacidad de ensayos en primates y de producción.