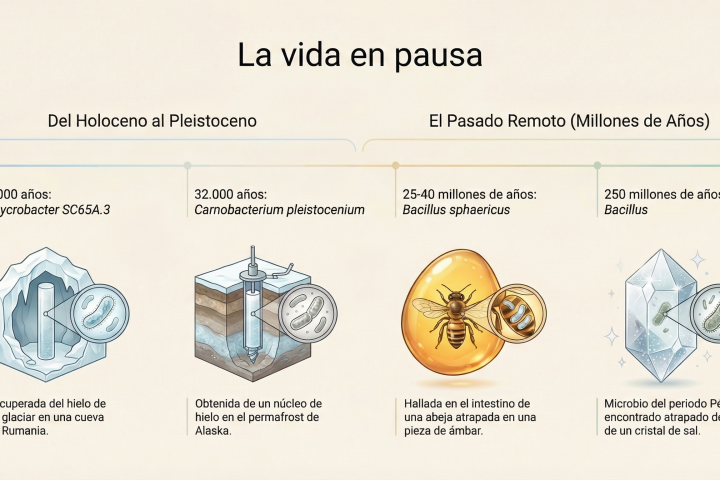
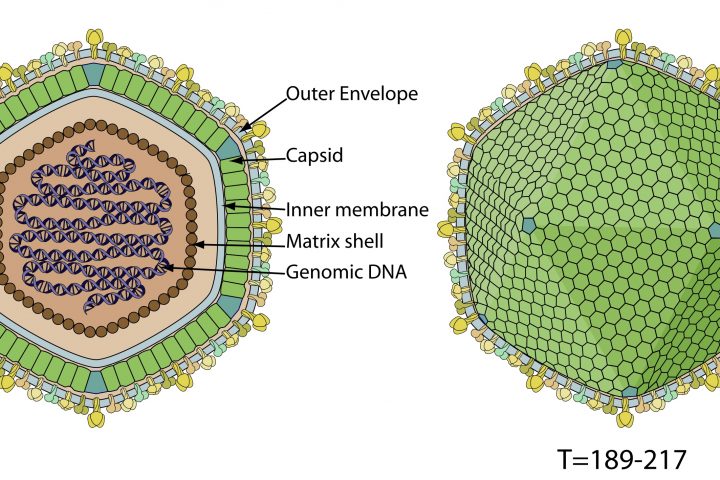
El pasado mes de agosto, los ganaderos
de la región de Renania del Norte en Alemania observaron una serie de síntomas
en su ganado: diarrea, fiebre, disminución de la producción de leche, inapetencia,
con una duración de 2 a
3 semanas. Alertaron del problema a las autoridades competentes y comenzó así una
investigación en el Friedrich Loeffler Institute en busca de la causa de esta posible enfermedad.
En primer lugar se descartaron los
virus comunes en el ganado que causan síntomas similares como los de la fiebre
aftosa, lengua azul, herpes,… Puesto que no era ninguno de ellos, decidieron
realizar un estudio metagenómico que
consiste en la secuenciación masiva de todos los genomas presentes en la
muestra y posterior análisis bioinformático. Para ello, tomaron tres muestras
de sangre de animales enfermos y secuenciaron todo el ADN presente. Detectaron
miles de secuencias de genomas de eucariotas, bacterias y virus. Tras el
análisis bioinformático concluyeron la presencia de un nuevo virus perteneciente
al grupo de los Bunyavirus, que denominaron provisionalmente virus Schmallenberg,
por ser el nombre de la granja de donde procede la primera muestra que dio
positivo para este nuevo virus.
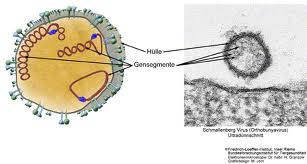
Los Bunyavirus son virus con
envoltura y genoma del tipo ARN monocatenario de sentido negativo, dividido en
tres segmentos: S, M y L.
Los Bunyavirus se transmiten por picaduras de
insectos (artrópodos),
y por eso se incluyen dentro de los arbovirus (arthropod borne virus).
Recientemente se han obtenido las
primeras imágenes de microscopía electrónica del virus Schmallenberg.
Los investigadores han desarrollado
un método de detección específico para este nuevo virus basado en una técnica
de amplificación génica por PCR (RT-qPCR). Mediante este test se ha detectado
la presencia del virus en el ganado (ovejas, vacas y cabras) también en los
Países Bajos, Bélgica, Francia, Inglaterra, Italia, Luxemburgo y recientemente en
España (el 14 de marzo en Córdoba), y ya ha sido reconocido oficialmente por
las autoridades de la Comisión Europea.
Esta distribución del virus no quiere
decir que la infección necesariamente comenzó en Alemania y se ha ido extendiendo
por toda Europa, sino que lo más probable es que el virus se ha descubierto por primera
vez allí y al aplicar la nueva tecnología de detección se comprueba que está
distribuido por muchos países y es más común de lo que se podría creer.
Respecto a la transmisión del virus,
se ha visto que puede ocurrir a través de la picadura de mosquitos, así como
por vía transplacentaria. Debido al aumento de estos insectos durante los meses
de verano y otoño, se espera una mayor transmisión en esa época, aunque
dependerá de la temperatura, ya que por debajo de 10-15º C el virus es inactivo.
Aunque su primera detección ha sido
por técnicas genómicas, el virus se ha podido aislar y multiplicar en cultivo
celular. Además, siguiendo los clásicos postulados de Koch, la enfermedad se ha reproducido al inocular animales sanos con el
virus. Se espera obtener una vacuna en los próximos dos años. Hasta el momento
no se ha producido ninguna enfermedad en humanos debida a este virus. Otros
virus genéticamente muy similares no han causado enfermedad humana, por lo que
es poco probable que el virus Schmallenberg la cause. Sin embargo, no se puede
excluir totalmente esta posibilidad, por lo que se ha recomendado tomar precauciones a las
personas que estén en contacto con el ganado.
El caso del virus Schmallenberg es
un ejemplo de la posibilidad de emplear la secuenciación directa de los genomas
para detectar patógenos emergentes, sin necesidad de cultivarlos. Un ejemplo de
que la metagenómica supone un cambio de rumbo de la microbiología.
Este post ha sido realizado por María
Villalba,
Aldara Freitas e Irati Garmendia, alumnas de la asignatura de Virologíade la Facultad de Ciencias
de
la Universidad de Navarra.
Novel
Orthobunyavirus in cattle, Europe, 2001. Hoffmann, B., et al. Emerg Infect Dis 2012,
18 (3).