Encuentran virus en
heces fosilizadas de la Edad Media
En 1996, durante unas excavaciones arqueológicas en la
ciudad belga de Namur, se encontró un barril sellado intacto de la Edad Media,
siglo XIV, de hace más de 500 años!. El barril estaba enterrado debajo de una
plaza a casi cuatro metros de profundidad, y ha estado protegido de la
contaminación ambiental durante siglos. ¿Qué tesoros de valor incalculable
contenía el barril?: coprolitos o lo que es lo mismo caca humana fosilizada (gr. kopros, excremento y lithos,
piedra). Se trataba de un barril que se empleaba en aquella época como
letrina, perfectamente cerrado y que había permitido la fosilización del
material fecal.

De momento se desconoce el autor de semejante “tesoro”, que
no ha sido todavía reclamado por ninguna autoridad o gobierno. El
descubrimiento quizá no llegue a exponerse en ningún museo, pero un grupo de
microbiólogos franceses (los microbiólogos somos gente muy seria) pensaron que
el análisis de esos coprolitos podría dar información sobre los microbios intestinales
y ambientales de la Edad Media. En concreto, han estudiado la diversidad de
virus en la muestra fosilizada.
Para evitar posteriores contaminaciones, los investigadores
cuidaron especialmente las condiciones de asepsia y sólo emplearon para sus análisis
la parte más interna del coprolito. Han demostrado la presencia de virus de
tres formas distintas, mediante microscopía electrónica, por secuenciación
masiva del DNA o metagenómica y por amplificación de secuencias específica
mediante PCR. Estas técnicas no requieren el cultivo de los virus, ni siquiera
conocer de antemano la secuencia de su genoma y permiten identificar nuevos
virus.
Han purificado las partículas virales, teñido y observado
mediante un microscopio electrónico. Las imágenes obtenidas demostraron estructuras
muy similares a virus (VLP, virus-like
particles) de unos 100-250 nm, algunos rodeados de una estructura parecida
a la envoltura y muchos de ellos muy similares a los bacteriófagos (virus que
infectan bacterias) típicos con cabeza y cola del grupo de los Siphovirus.
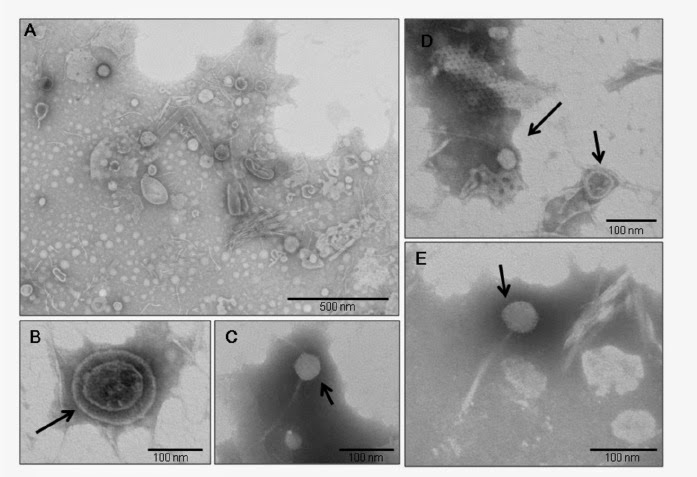
Imágenes de microscopía electrónica de partículas virales
aisladas del coprolito. En (B) una partícula similar a un virus de unos 100 nm,
rodeada de una estructura similar a una envoltura viral. En (C) y (E)
partículas con cabeza icosaédrica y cola filamentosa típica de los
bacteriófagos del grupo de los Siphovirus,
como el virus Lambda.
Además, han purificado el DNA de las muestras, han secuenciado,
procesado y ensamblado las secuencias y comparado con las bases de datos. Más
del 85% de las secuencias eran similares a virus DNA, la mayoría del tipo de
los bacteriófagos de las familias Siphoviridae
(59%), Myoviridae (9%) y Podoviridae (6%). Pero también encontraron
secuencias similares a virus de eucariotas
(Poxvirus, Adenovirus, Mimivirus,
Herpes, Baculovirus, …) y algunos de arqueas.
La mayoría de las secuencias similares a bacteriófagos
mostraron homología con fagos de Bacillus,
pero se encontraron bacteriófagos que infectan a más de 37 géneros de bacterias
diferentes: virus que infectan a enterobacterias, lactobacillus, lactococcus, bacterias
del suelo y algunos de patógenos humanos, como Mycobacterium, Staphylococcus,
Pseudomonas, Listeria, …
Han demostrado además que algunos eran bacteriófagos lisogénicos,
es decir, capaces de integrar su genoma en el de la bacteria huésped en forma
de profagos. Muchos de los fagos lisogénicos llevan en su genoma secuencias del
genoma de la bacteria. Por eso, también han amplificado mediante PCR secuencias
concretas para confirmar algunos de estos datos de secuenciación. Buscaron
mediante PCR secuencias de genes de virulencia bacteriana entre las secuencias
de estos fagos. Encontraron genes de resistencia al antibiótico cloranfenicol y
factores de virulencia. Esto confirma que los fagos son un reservorios o
almacén de genes de resistencia a antibióticos. Confirma que la evolución y
dispersión de estos genes de resistencia comenzó mucho antes del uso de los
antibióticos: los antibióticos son sustancias naturales producidas por las
bacterias y hongos principalmente del suelo y la evolución y diseminación de
los genes de resistencia a los antibióticos comenzó mucho antes de su uso por
el ser humano.
Al comparar los virus del coprolito fosilizado con los de
muestras (heces) humanas modernas demostraron que el conjunto de virus del
coprolito de la Edad Media es funcionalmente más diverso y más rico en genes de
virulencia bacteriana que los actuales.
La cantidad de información que se puede sacar de un
coprolito fosilizado!
Appelt, S., et al. (2014). Viruses in a 14th-Century Coprolite Applied and Environmental Microbiology, 80 (9), 2648-2655 DOI: 10.1128/AEM.03242-13