El mayor proyecto de secuenciación de genomas completos bacterianos
Sí, ya sé que el título es bastante sensacionalista, la
bacteria “come carne”. Pero así es como se llamó al grupo A de Streptococcus
pyogenes, una bacteria Gram positiva, que causa miles de infecciones invasivas muy graves. Esta
bacteria produce fascitis necrotizante (gangrena
estreptocócica), una infección aguda que se extiende por el tejido subcutáneo y
produce una rápida destrucción y necrosis de los tejidos (por razones de buen
gusto, no incluyo ninguna imagen de casos de fascitis necrotizante). La mortalidad puede llegar a casi el 50%,
aunque depende de si el diagnóstico y el tratamiento antibiótico y/o quirúrgico son rápidos y adecuados.Se calcula que esta bacteria es responsable de más de 600
millones de infecciones en todo el mundo cada año y entre 10.000 y 15.000 casos
muy graves sólo en EE.UU.
Después de varias décadas en las que disminuyó la incidencia
de esta enfermedad, a finales de los 80 y principios de los 90, de forma
sorprendente resurgieron en muchos países varios brotes de infecciones invasivas muy graves porel serotipo Emm proteína
1 (M1) de Streptococcus pyogenes
grupo A. Algunas de estas infecciones fueron muy mediáticas porque afectaron
incluso a personajes muy populares, como Jim
Henson, el creador de los teleñecos, que murió por una infección por esta
bacteria.

James Maury «Jim» Henson fue productor televisivo
estadounidense, conocido por ser el creador de The Muppets, los teleñecos. Falleció el 16 de mayo de 1990 a los 53
años de edad, por una infección por Streptococcus
pyogenes grupo A, unas 20 horas después de llegar por su propio pie a
emergencias del hospital de Nueva York, sin ser consciente de lo grave que
estaba.
Hasta ahora ha sido todo un misterio cómo surgió esta cepa concreta tan virulenta. Para descubrirlo, un
grupo de investigadores han secuenciado un total de 3.615 genomas completos de
aislamientos de la bacteria obtenidos desde 1920 hasta 2013 en ocho países distintos
de Europa y América (Dinamarca, Alemania, Finlandia, Islandia, Noruega, Suecia,
Estados Unidos y Canadá).
El objetivo de este trabajo, hasta el día de hoy el mayor proyecto de secuenciación de
genomas completos bacterianos, es descubrir cómo han sido los eventos
moleculares que han contribuido a la aparición, diseminación y diversificación
genética de este clon tan virulento que ha llegado a causar infecciones humanas
tan graves por todo el planeta.
Streptococcus pyogenes
grupo A es una bacteria Gram positiva con forma de coco que crece en cadenas.
Expresa en su pared celular el antígeno grupo A de la clasificación de
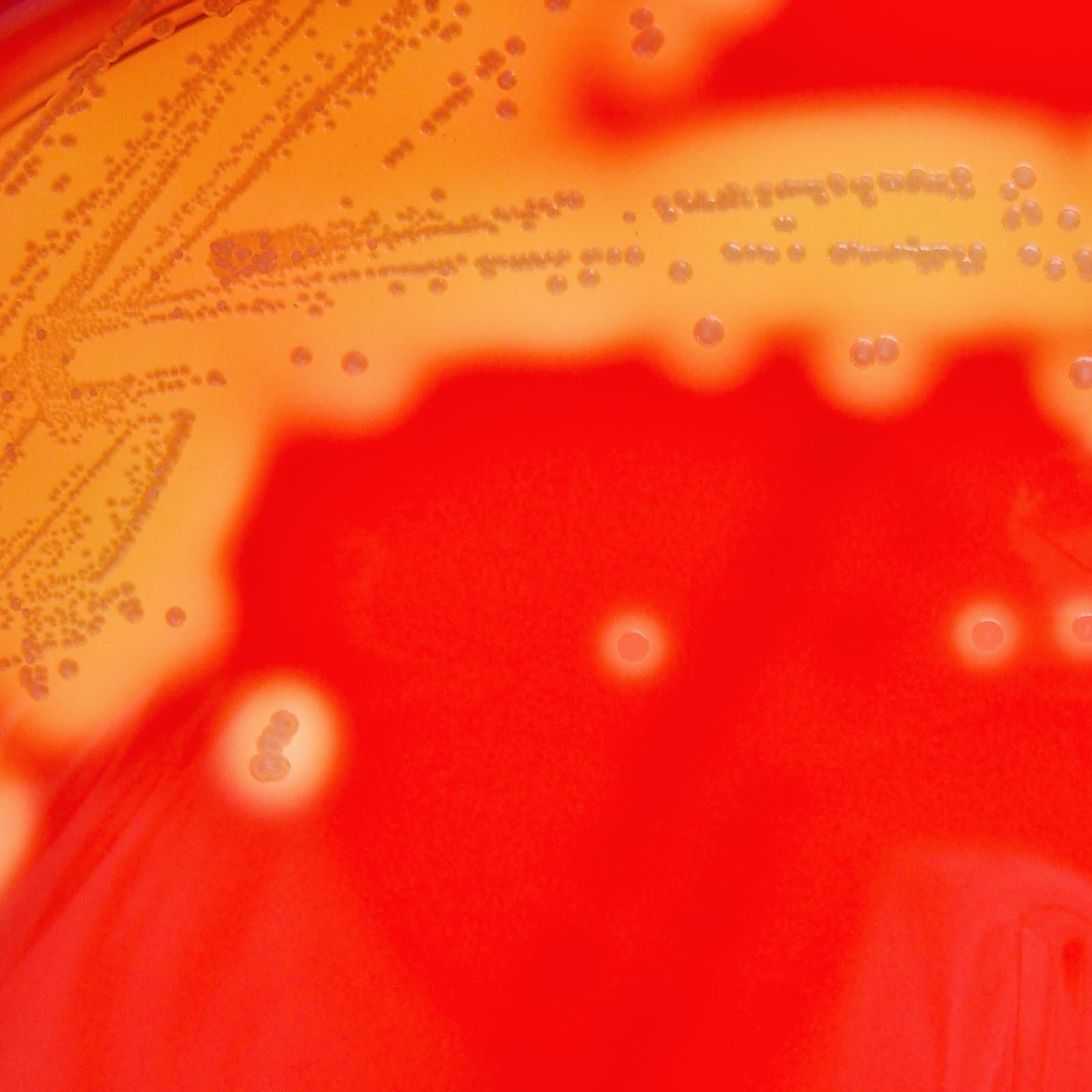
Lancefield y produce hemólisis del tipo beta (grandes halos de hemólisis)
cuando se cultiva en agar sangre. Se le conoce también por estreptococo
beta-hemolítico del grupo A (o por sus siglas en inglés GAS, Group A Streptococcus).
Es la causa más frecuente de faringitis bacteriana y de escarlatina, una
complicación de la faringitis que se produce cuando la bacteria contiene un
fago que estimula la producción de una toxina.
Además, Streptococcus
pyogenes grupo A es un buen modelo
para hacer este tipo de estudios porque produce muchas infecciones humanas, hay
abundantes cepas recolectadas de los últimos 90 años y de dos continentes
distintos, su genoma es relativamente pequeño (1,8 Mb) y fácil de secuenciar, y
es un patógeno sólo humano, no de animales, el hombre es el único huésped.
Los datos sugieren que el actual clon epidémico surgió a partir
de una bacteria precursora, paso a paso, adquiriendo de manera secuencial
distintos elementos genéticos que la hicieron mucho más virulenta. Primero incorporó
a su genoma un bacteriófago (un
virus que infecta bacterias) con los genes del factor de virulencia de la DNAsa
extracelular (SdaD2). Posteriormente adquirió otro bacteriófago con
los genes SpeA1, una variante de la
exotoxina superantígeno A. Luego, este mismo gen evolucionó a una nueva
variante, SpeA2, por un fenómeno de mutaciónpuntual, antes de adquirir por transferencia
genética horizontal una región grande de 36 Kb de DNA cromosomal que
contiene los factores de virulencia de la glicohidrolasa
y la streptolysina O. La adquisición de este fragmento grande debió
de ocurrir a principios de los 80 (probablemente en 1983) en una bacteria que ya contenía los genes SdaD2 y SpeA2. A
partir de ahí, este clon se extendió rápidamente por todo el mundo, dando lugar
a la cepa actual mucho más virulenta y que ha causado las epidemias explosivas durante
los últimos 25 años.
Este trabajo, demuestra los
eventos moleculares que han ocurrido paso a paso y que han hecho que de una
única célula bacteriana acaben produciéndose millones de infecciones por todo
el mundo.
La secuencia de eventos
moleculares más probable, por tanto, ha debido ser:
– adquisición del gen SdaD2 (DNAsa extracelular)
de un fago
– adquisición del gen SpeA1 (exotoxina del
superantígeno A) de otro fago
– mutación puntual de SpeA1 para generar la
variante SpeA2
– adquisición por transferencia horizontal de los
genes glicohidrolasa y streptolisina O en un fragmento de 36 Kb
Este tipo de estudios son esenciales para mejorar las
estrategias para reconocer y predecir la aparición de nuevas cepas virulentas y
epidemias, ayudan a formular políticas de protección de salud pública y a
desarrollar nuevas vacunas. En definitiva, en este caso se trata de “comerle el
terreno” a este bicho tan desagradable.
Evolutionary pathway to increased virulence and epidemic
group A Streptococcus disease derived from 3,615 genome sequences. 2014.
Nasser, W., et al. Proc Natl Acad Sci U S A. 111(17):E1768-76. doi: 10.1073/pnas.1403138111.