Las nuevas técnicas de análisis genómicos permiten descubrir cómo eran,
qué comían e incluso qué microbios tenían nuestros antepasados
Reconstruir un dinosaurio completo a partir de unos trocitos
de DNA atrapado en un fósil de ámbar, como nos hicieron creer el Jurassic Park,
es imposible … de momento.

Hasta hace unos pocos años, el DNA antiguo que quedaba
preservado en restos arqueológicos era muy difícil de analizar. Estaba muy
dañado y poca información se podía sacar de él. Sin embargo, hoy en día hay nuevas
técnicas que permiten amplificar, secuenciar y analizar el genoma antiguo y que
están revolucionando nuestro conocimiento sobre la prehistoria de la humanidad.
Muchas veces este trabajo depende de la disponibilidad de muestras apropiadas. No
somos capaces de revivir un dinosaurio, pero veamos tres ejemplos de lo que hoy
en día podemos hacer con el DNA antiguo: ¡vas a alucinar!
Los secretos
escondidos en un “chicle” de la Edad de Piedra
En diciembre de 2019 se publicó en la revista Nature Communications (1) un interesante
trabajo en el que describían cómo había logrado secuenciar el genoma humano completo y el microbioma de la boca de un individuo
que vivió hace 5.700 años, a partir de una goma de resina de abedul. Esta
goma había sido usada como si fuera un chicle y conservaba el DNA de la persona
que lo mascaba y permitió incluso identificar las bacterias de su boca y lo que
había comido antes de masticarlo.

En realidad la muestra era una especie de bola oscura de
brea o resina que se producía al calentar la corteza de abedul, que se usaba
entonces para pegar herramientas de piedra. La presencia de marcas de dientes
sugiere que la sustancia fue masticada, quizás para hacerla más maleable o incluso
para aliviar el dolor. Estaba enterrada bajo una capa de lodo, lo que ha
ayudado a su preservación, y provenía de un yacimiento de la Edad de Piedra en
Saltholm, una isla danesa en el mar Báltico.
Es la primera vez que se extrae un genoma humano antiguo completo de
otro material que no sea un hueso
De esta muestra han podido extraer suficiente DNA humano
como para secuenciar el genoma completo del individuo. Hoy las técnicas de
genómica forense permiten detectar determinados genes o mutaciones que
proporcionan mucha información sobre las características del individuo del que
proviene ese DNA. Así, el análisis de este genoma antiguo ha permitido desvelar
que se trataba de una mujer,
genéticamente vinculada con los cazadores-recolectores de la Europa continental.
Probablemente descendía de una población de colonos que se trasladó desde
Europa occidental, después de que se retiraran de los glaciares. Además, han
podido determinar que tenía la piel
oscura, el cabello castaño oscuro
y los ojos azules. Aunque esto no
está en los genes, los investigadores la han puesto nombre: Lola. Saben incluso
que Lola era intolerante a la lactosa.
En el genoma no encontraron la mutación que permite a la mayor parte de los
humanos modernos beber leche animal sin indigestarse.

Recreación artística del aspecto que pudo tener Lola (Tom Björklund)
Los investigadores también pudieron extraer DNA de los microbios
atrapados en el «chicle». El análisis demostró que la mayoría
pertenecía a microbios beneficiosos de
la microbiota oral humana. Pero también detectaron DNA de algunas bacterias
patógenas como Porphyromonas gingivalis y Treponema
denticola que causan infecciones
en las encías y periodontitis, y el virus Epstein-Barr, uno de los más
comunes y que actualmente infecta al 90% de la población humana adulta. De las
secuencias de DNA que obtuvieron fueron capaces también de reconstruir el genoma completo de Streptococcus
pneumoniae, una bacteria que
puede causar neumonía. Sin embargo, los investigadores afirman que los datos no
permiten saber el estado de salud de Lola.
Pero además, los investigadores hallaron restos de DNA que
no eran ni humanos ni bacterianos: unos eran de origen animal, de un ánade o
pato real; y otros de procedencia vegetal, de avellanas. De ahí, que deduzcan
que Lola debió de comer pato con avellanas poco antes de mascar el “chicle” de
abedul.
Los microbios de los
neandertales
Pero estas técnicas no solo nos permiten conocer la historia
de nuestros antepasados de la Edad de Piedra, sino irnos incluso mucho más
lejos, hasta los neandertales, con los que convivimos durante unos miles de
años. Habitaron Europa y Asia Occidental desde hace aproximadamente 200.000
años, hasta que definitivamente se extinguieron hace unos 28.000 años. Eran
principalmente cazadores y solían vivir en pequeños grupos de unos 15-30
individuos. Convivieron con los Homo
sapiens durante el Pleistoceno y, según los últimos datos genómicos, en
nuestro genoma actual hay restos de DNA neandertal, lo que demuestra que nos
cruzamos, en el sentido sexual de la palabra, en algún momento de la
prehistoria.
Pero los neandertales se extinguieron y solo nos han llegado
hasta nuestros días unos pocos huesos. El registro fósil de los neandertales
está representado por unos 400 individuos. Obviamente de sus microbios no
sabemos nada … o casi nada.
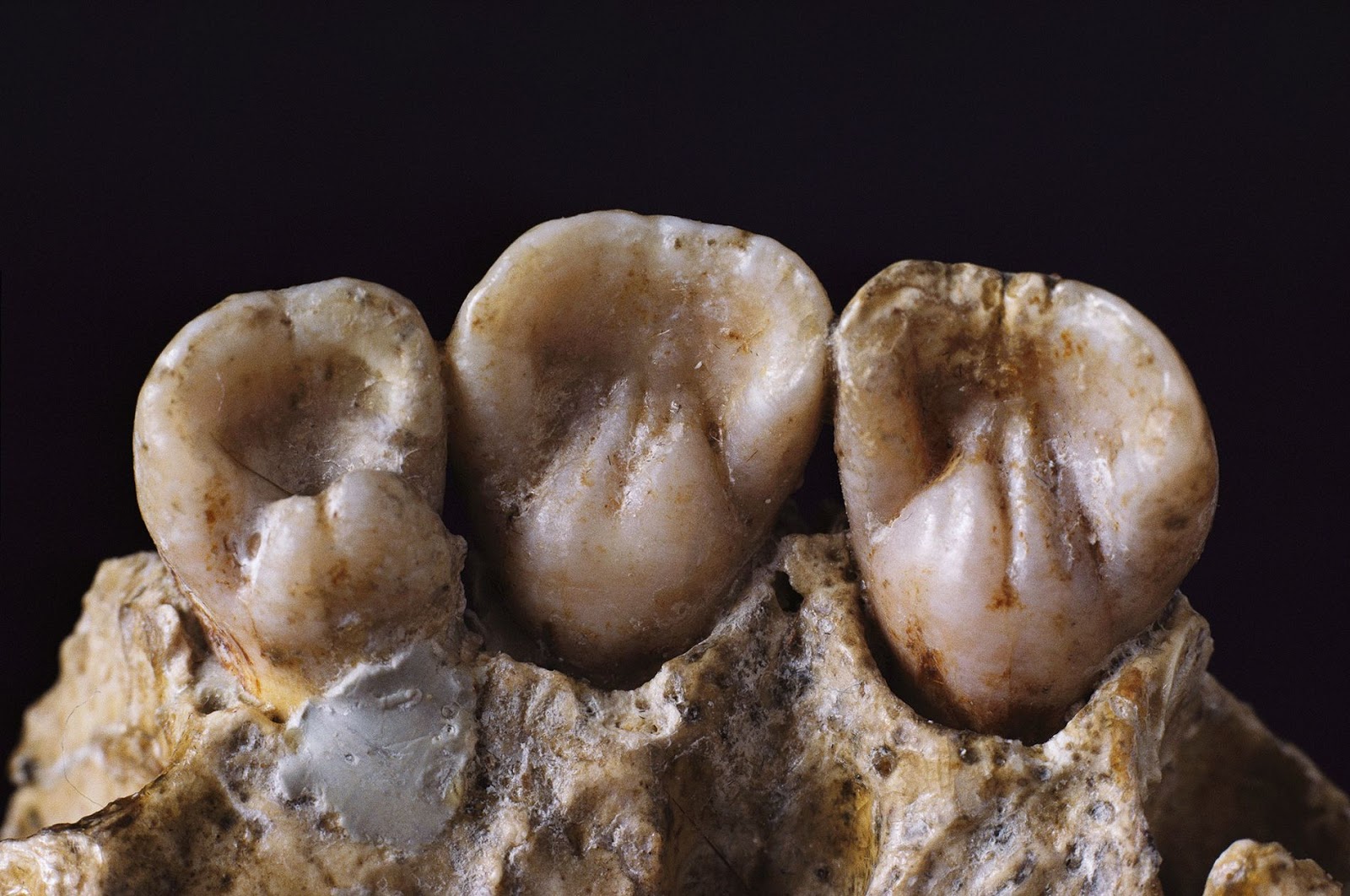
Hace unos pocos años, en 2017, se publicó un trabajo(2) en
el que descubrieron que al estudiar algunos huesos de la dentadura de los neandertales,
en las caries dentales, quedaba preservado DNA microbiano y que su análisis
podría darnos mucha información sobre cómo era la microbiota de nuestros
antepasados. Así que extrajeron el DNA de las caries, y lo secuenciaron. Emplearon
muestras de caries de cinco neandertales,
dos de la cueva de El Sidrón en España, dos belgas y un italiano.
Comprobaron que el 93% de las secuencias eran bacterianas,
el 6% de arqueas y el resto de microorganismos eucariotas y virus. Fueron
capaces de caracterizar hasta 222
especies de bacterias y los grupos bacterianos más frecuentes eran
similares a los que nos podemos encontrar en la placa dental de humanos
modernos: Actinobacterias, Firmicutes, Bacteroidetes, Fusobacterias,
Proteobacterias y Espiroquetas. Obviamente también encontraron secuencias de
bacterias que producen caries y otras enfermedades dentales, como Streptococcus mutans. Un dato
interesante es que fueron incluso capaces de secuenciar el genoma casi completo de una de las bacterias del
neandertal, que han denominado Methanobrevibacter
oralis subsp. neandertalensis, o sea una arquea simbionte que produce
metano encontrada en la boca de un neandertal. Han podido incluso estimar su
antigüedad en unos 48.000 años.
Methanobrevibacter oralis subsp. neandertalensis es el genoma
microbiano más antiguo hasta ahora secuenciado
Además, con los datos del DNA preservado en sus dientes, los
científicos fueron capaces de determinar que la dieta de los neandertales belgas era a base de carne de rinocerontes
lanudos y muflones —un tipo de cabra salvaje europea—, mientras que la de los
españoles era vegetariana, a base de champiñones, musgos y piñones —todavía no
habían inventado ni la cerveza belga ni la paella—. En los dientes de los
neandertales de El Sidrón también han encontrado secuencias de DNA del hongo Penicillium,
que produce antibióticos. Los autores lo han interpretado como que ya nuestros
antepasados se medicaban miles de años antes del descubrimiento de los
antibióticos, pero teniendo en cuenta que la cueva de El Sidrón está en
Asturias, bien pudiera ser que ya comían queso de Cabrales prehistórico (je,
je).
Los investigadores también examinaron la diversidad
microbiana en las muestras de los neandertales en busca de potenciales microorganismos patógenos que fueran un
signo de enfermedad. Encontraron secuencias de un microorganismo eucariota
patógeno —Enterocytozoon bieneusi—
que infecta las células del epitelio intestinal y produce diarreas. Así que se
demuestra que los neandertales… tenían diarrea. También encontraron que la
microbiota neandertal contenía menos bacterias Gram-negativas potencialmente
patógenas, que son más frecuentes en los humanos modernos. Pero sí detectaron
especies potencialmente patógenos como Neisseria
gonorrhoeae, Corynebacterium diphteriae o Bordetella parapertussis, aunque no es posible asegurar si estas
secuencias son en realidad de cepas similares no patógenas. Así que no podemos
afirmar con seguridad que los neandertales padecieran gonorrea, difteria o
tosferina, pero sí que tenían caries y diarrea…, y que producían metano.
El rostro de los
denisovanos en un meñique
Pero quizá lo más alucinante de todas estas técnicas de
análisis de DNA antiguo lo hemos conocido hace unos pocos meses. En septiembre
de 2019 se publicó un artículo (3) en el que mostraban la reconstrucción del aspecto
físico de un tipo de homínido gracias a un novedoso método a partir del
análisis genómico de la falange de un meñique.
El homínido en
cuestión eran un denisovano, un grupo de homínidos cuya historia es
apasionantes. Se trata de unos antepasados nuestros que vivieron en Siberia y
Asia oriental hace más de 50.000 años y que son un misterio para los
científicos. Se descubrieron hace solo una década en las cuevas de
Denísova en los montes Altái de Siberia. De los denisovanos solo se han
encontrado cuatros trocitos fosilizados: una falange de dedo meñique,
tres dientes y una mandíbula inferior. Nada más. Pero a partir de ahí, se ha
podido extraer, secuenciar y analizar el genoma de los denisovanos. Al
contrario de lo que ocurre con el resto de especies humanas que han sido identificadas
gracias a sus fósiles, de los denisovanos todo lo que conocemos lo sabemos a partir
del análisis de su DNA.
Se ha sugerido que este homínido vivió entre hace un millón
de años y 40.000 años, en áreas en las que también vivían neandertales y Homo sapiens. Su origen se encontraría
en una migración de África distinta de las asociadas con humanos modernos y
neandertales. El análisis del DNA indica que debió de existir un ancestro común
entre denisovanos, neandertales y el Homo
sapiens.
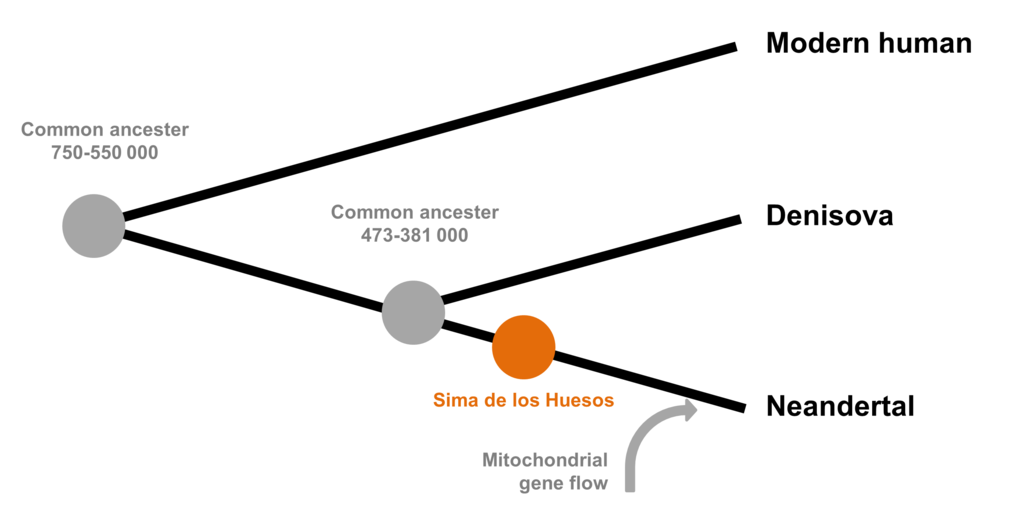
Y lo mismo que ocurrió con los neandertales, los denisovanos
también se cruzaron con los sapiens
que vivía en Eurasia en ese momento. No solo eso sino que las pruebas genéticas
demuestran que uno de los restos de denisovanos tiene cromosomas que heredó de
una madre neandertal y de un padre denisovano. Es decir, se trata de un
descendiente híbrido directo de dos “especies” humanas distintas y ahora
extintas.
Parece mentira que todo esto lo sepamos del análisis del DNA
extraído de la falange de un meñique
fosilizado. Pero todavía nos queda lo mejor.
Como hemos comentado, en septiembre de 2019 se publicó un
artículo en el que mostraban la
reconstrucción del aspecto físico de un denisovano a partir del análisis
genómico del huesecillo del meñique. Para ello, los investigadores han aplicado
una nueva técnica de
análisis genómico que permite descubrir los patrones de metilación (un tipo
de modificación química del DNA) que se relacionan con la expresión de
determinados genes.
La metilación del DNA te permite saber qué genes están “encendidos” o
“apagados”
Así se ha podido asociar cambios en la actividad de los
genes con cambios anatómicos para predecir así la apariencia física. Se
identificaron un centenar de genes silenciados en los denisovanos que, por el
contrario, sí estaban activos en sapiens y neandertales. Estos datos se
cruzaron con la información recogida en enormes bases de datos de enfermedades
monogénicas (causadas por un único gen) que provocan un determinado desarrollo
morfológico. Tras realizar el estudio genético, pudieron determinar algunas
características anatómicas de nuestros parientes y realizar un diseño de su cabeza y cara mediante programas
informáticos.
Para comprobar la eficacia del método, los investigadores primero demostraron que su técnica era capaz de reconstruir
con precisión la anatomía ya conocida de los neandertales y los chimpancés.
Los resultados fueron comparados con un esqueleto de neandertal y los rasgos
predichos coincidían entre el 85% y el 90%. En el caso de los chimpancés, el
acierto se incrementaba hasta el 92%.
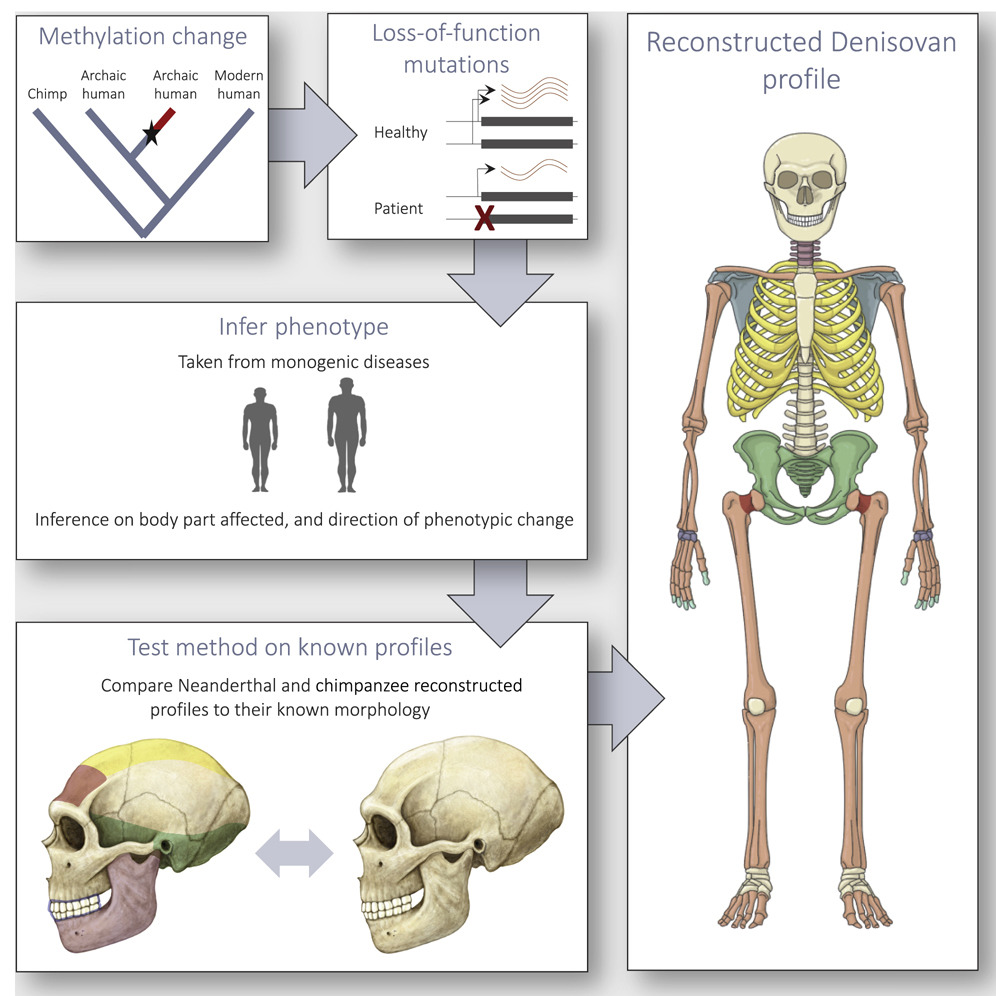
Modelo anatómico de un
denisovano (Fuente: ref 3)
Así, han podido estimar hasta 56 rasgos que caracterizan la fisionomía de los denisovanos, 34 de ellos del
cráneo. Solo once son específicos de los denisovanos, mientras que el resto son
compartidos con neandertales o humanos modernos.
En muchos rasgos se parecían a los neandertales, por
ejemplo, en su frente inclinada, la cara alargada y la pelvis grande. Sin
embargo, en otros rasgos resultan diferentes, como su gran arco dental, su cráneo mucho
más ancho que el nuestro o el de
los neandertales, y el que no tenían mentón. En general, los denisovanos resultaban
ser más altos y fuertes que los neandertales. Este trabajo demuestra que el
estudio de la metilación del DNA puede emplearse para reconstruir
características anatómicas de las que incluso no haya registro fósil.

Así debían ser los denisovanos, según el análisis del DNA de uno de sus meñiques
Estos tres ejemplos, la fisionomía del rostro de los
denisovanos, los microbios que tenían los neandertales o cómo eran nuestro
antepasados de la Edad de Piedra, demuestran de lo que somos capaces de hacer
hoy en día con las técnicas genómicas a partir de un huesecillo de un meñique,
de una caries fosilizada o de un “chicle” de hace miles de años. Lo de Jurassic
Park, ¿seguirá siendo ciencia ficción?
Referencias:
(1) A 5700 Year-Old Human Genome and Oral Microbiome From Chewed Birch Pitch. Jensen, T.Z.T., y col. Nat Commun 2019. 10 (1): 5520.
(2) Neanderthal Behaviour, Diet, and Disease Inferred From Ancient DNA in Dental Calculus. Weyrich, L. S., y col. Nature. 2017. 544
(7650), 357-361.
(3) Reconstructing Denisovan Anatomy Using DNA Methylation Maps. Gokhman, D., y col. Cell. 2019. 79 (1), 180-192.







Realmente fascinante