¿Qué pasa si analizas el genoma de
una bacteria después de 50.000 generaciones?
Uno de los debates más intensos entre los
evolucionistas del siglo XX se centró en conocer si la fuerza que gobierna la
evolución de los diferentes genomas es la selección
o el azar. La controversia entre
seleccionistas y neutralistas surgió a finales de los 1960s cuando el japonés
Motoo Kimura propuso la teoría
neutralista de la evolución molecular para explicar la observación de que
los cambios en la secuencia de algunas proteínas entre diversas especies
parecían producirse de manera constante y proporcional a su tiempo de
divergencia y a una tasa muy superior a la que podría justificarse si sólo se
produjeran por selección.
La teoría neutralista de la evolución molecular
explica por qué hay tantos cambios en la secuencia de un mismo gen entre los
diferentes organismos y su aparición de manera constante en el tiempo. Esto
permite establecer un reloj molecular y cuantificar el tiempo de divergencia
entre ellos.
Los individuos que conforman la siguiente generación
son descendientes de los individuos que tuvieron hijos y el pensamiento
darwinista tiende a pensar que los individuos mejores o más adaptados son los
que dejan un mayor número de descendientes. Esto haría que la evolución o
cambio de una especie esté guiada por la selección
natural. Sin embargo, esto no tiene porqué ser siempre así. Algunas veces
el azar juega un papel importante.
Los individuos que tienen descendientes no tienen por qué ser los mejor adaptados
sino que ha podido ocurrir así por cuestiones que pueden no tener que ver con
sus características biológicas (por lo que desde el punto de vista biológico
estaría actuando el azar). Esto es especialmente importante en poblaciones
pequeñas en un fenómeno que se conoce como deriva
genética.
La mutación
es un fenómeno que se produce a una tasa más o menos constante en condiciones
normales. Esta mutación podrá ser beneficiosa, perjudicial o neutral (ni
beneficiosa ni perjudicial) con respecto a la aptitud biológica del individuo
que la porta. Si es perjudicial desaparecerá en una o varias generaciones ya
que los individuos que la tienen serán menos aptos. Por el contrario, si es
beneficiosa su frecuencia irá aumentando en la población ya que los individuos
que la portan tendrán más descendientes y éstos serán más viables. Ambos tipos
de cambios conducen a una evolución
adaptativa al ambiente en el que vive el organismo.

Pero, ¿qué ocurre con las mutaciones neutrales? Su frecuencia aumentará o disminuirá dependiendo
del azar; algunas se perderán, pero otras aumentarán en frecuencia llevando a
una evolución NO adaptativa. Hay
cambio, pero los individuos no están ni más ni menos adaptados. La teoría
neutralista de Kimura se basa en que este tipo de mutaciones son las más
frecuentes en una población; no implica que la selección no actúe sino que ésta
lo hace frente a una proporción muy pequeña de cambios moleculares, los
beneficiosos o los perjudiciales. La mayor parte de los cambios a nivel
molecular son neutrales, no hacen mejor o peor al individuo que los porta. Por
ello se acumulan de manera constante en el tiempo permitiendo establecer un reloj molecular en la evolución de los
distintos linajes.
Hay dos tipos de evolución: la adaptativa y la NO
adaptativa
Existen múltiples ejemplos experimentales que muestran
la existencia de mutaciones beneficiosas que son muy frecuentes en algunas
poblaciones frente a otras de la misma especie, como la permanencia de la
actividad lactasa en algunos grupos humanos. Sin embargo, a nivel general, se
desconoce cuántas de las nuevas mutaciones que se van produciendo son
beneficiosas. Conocer esta tasa puede ser útil para mejorar nuestro
conocimiento de la evolución molecular y los métodos que ayuden a datar y
reconstruir la historia evolutiva.
La secuenciación
rápida de genomas completos está facilitando la realización de ensayos
experimentales que nos ayuden a profundizar en ello. Sin embargo, a pesar de
que las mejoras técnicas facilitan estos estudios, hay algo imprescindible en
los análisis evolutivos: la paciencia,
ya que debemos analizar cientos o miles de generaciones para tener datos
fiables.
Keep calm and
be patient
Las bacterias son, de nuevo, nuestras mejores aliadas
para poder monitorizar tantas generaciones en un tiempo razonable. Hasta la fecha
algunos análisis se habían realizado siguiendo a varias decenas de bacterias
durante algunos cientos de generaciones o a un clon durante casi 40.000
generaciones. A pesar de ello era complicado conocer qué mutaciones son
realmente beneficiosas (o conductoras de la selección) y cuáles simplemente
acompañan a las beneficiosas (son pasajeras). Recientemente un estudio
publicado en Nature muestra los resultados del análisis del genoma completo de dos clones de 12
poblaciones de E. coli tras 500,
1.000, 1.500, 2.000, 5.000, 10.000, 15.000, 20.000, 30.000, 40.000 y 50.000
generaciones (¡desde hace 28 años!). Esto hace un total de 264 genomas
completos cuyo análisis ha mostrado algunos resultados curiosos y otros que apoyan
algunas ideas conocidas.
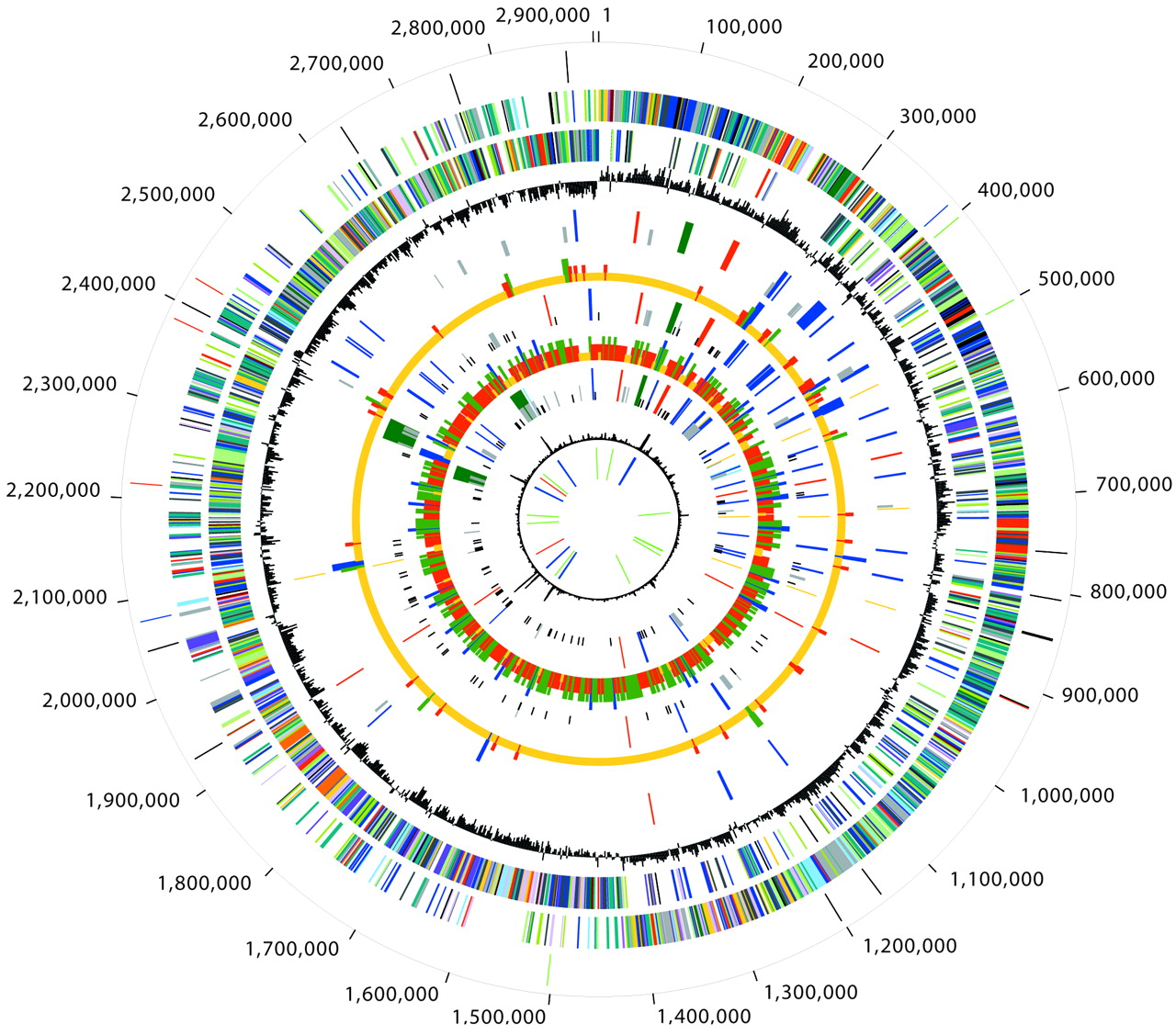
(Mapa del genoma de una bacteria)
¿Cómo cambia el
genoma de una bacteria después de 28 años (50.000 generaciones) multiplicándose?
De manera sorprendente la longitud media de los
genomas tras 50.000 generaciones había disminuido en casi un 1,5% desde la
bacteria ancestral y las mutaciones no se habían distribuido de manera uniforme
a lo largo de las 12 poblaciones analizadas. Seis de ellas habían evolucionado
hacia un fenotipo hipermutador, lo
que les hacía acumular algo más del 96% de todas las mutaciones detectadas. Sin
embargo esta “hipermutabilidad” disminuía con el tiempo, ya que parecía
favorecer la mayor presencia de mutaciones deletéreas o perjudiciales.
Si mutas mucho,
empiezas a cargarte cosas importantes
Además, este efecto se asocia a la presencia de un
mayor número de secuencias transponibles. Los transposones son secuencias presentes en la mayor parte de
organismos, entre los que se encuentra el nuestro, y que son capaces de saltar
de una posición a otra del genoma. Efectivamente desde hace años se conoce que
son responsables de una mayor inestabilidad genómica. Lógicamente la presencia
en un mayor o menor grado de este tipo de secuencias y el fenotipo hipermutador
puede cambiar el tiempo y modo de evolución del organismo que las porta. Aunque
estas bacterias teóricamente acumulaban una mayor tasa de mutaciones
beneficiosas también lo hacían de mutaciones deletéreas, compensándose ambos
hechos. Además en estas poblaciones se hacía más difícil diferenciar las
mutaciones realmente beneficiosas de las que no lo son en un “mar” de mutaciones.

Sin embargo, en las bacterias sin fenotipo
hipermutador se podía observar que las
mutaciones potencialmente beneficiosas se acumulaban a una tasa muy superior a
las neutrales. Sin embargo esta acumulación disminuía con el tiempo ya que
en las primeras 500 generaciones se acumulaban 17 veces más mutaciones
beneficiosas pero al acercarse a las 50.000 generaciones esta acumulación era
de sólo 2 veces más. Por otra parte, la mitad de estas mutaciones se
encontraban en sólo 57 genes (en sólo el 2% del genoma) que estarían favoreciendo
esta adaptación.
La adaptación,
al principio es muy rápida y luego disminuye. Pero la frecuencia de mutación neutral
se mantienen constante, de ahí que existe un reloj molecular.
Así, el color de la evolución no es ni blanco ni negro
sino gris (como casi todo). La mayor parte de las mutaciones fijadas parecen
ser beneficiosas, pero su proporción disminuye cuanto mayor es la adaptación.
Sin embargo las mutaciones neutrales se acumulan a una tasa bastante constante
en el tiempo lo que hace que el reloj molecular efectivamente exista.
Reloj
molecular: a más tiempo, más cambios
Por lo tanto a nivel molecular actúa tanto la
selección como el azar. En el fondo la evolución de los genomas se basan en continuos cambios en el equilibrio de
ambas fuerzas. En un primer momento en poblaciones grandes la selección
ganaría al azar, pero una vez conseguida una adaptación razonable el azar se
mantiene como una fuerza importante de los cambios que sufre un genoma.
El autor de esta entrada es José Luis Vizmanos, Catedrático
de Genética y profesor de Genética de poblaciones de la Universidad de Navarra.
(1) Tempo and mode of genome evolution in a 50,000-generation experiment. Tenaillon, O., et al. Nature 536, 165–170 (11
August 2016) doi:10.1038/nature18959







Se me ocurre un pero: Supongo (no he leído lo contrario) que durante esas 50.000 generaciones el medio se mantenía constante, lo que "alejaría" los resultados de la realidad.
Saludos.