El origen del SARS-Cov-2
VER ACTUALIZACIÓN SOBRE ESTE DEL 28 DE MAYO DE 2021
Desde hace unas semanas viene circulando por las redes sociales, e incluso por algunos medios de comunicación, la noticia de que el coronavirus SARS-Cov-2 es producto de un ensayo de biotecnología, ha sido creado en un laboratorio con fines perversos, y se ha escapado de un laboratorio de bioseguridad de Wuhan, … Según esta hipótesis conspiranoica, el origen del virus es artificial. Deberíamos recordar que exactamente las mismas teorías circularon cuando el VIH, el Ébola o el Zika. No somos muy originales.
La proteína S del virus interacciona con el receptor celular ACE2
Dejemos a un lado los mensajes de WhatsApp y vemos que nos dice la ciencia. De donde podemos obtener más información sobre el origen del virus es analizando su genoma. A día de hoy ya hay más de 2.600 genomas secuenciados de aislamientos obtenidos de 53 países (ver NextStrain). La cantidad de información es inmensa.
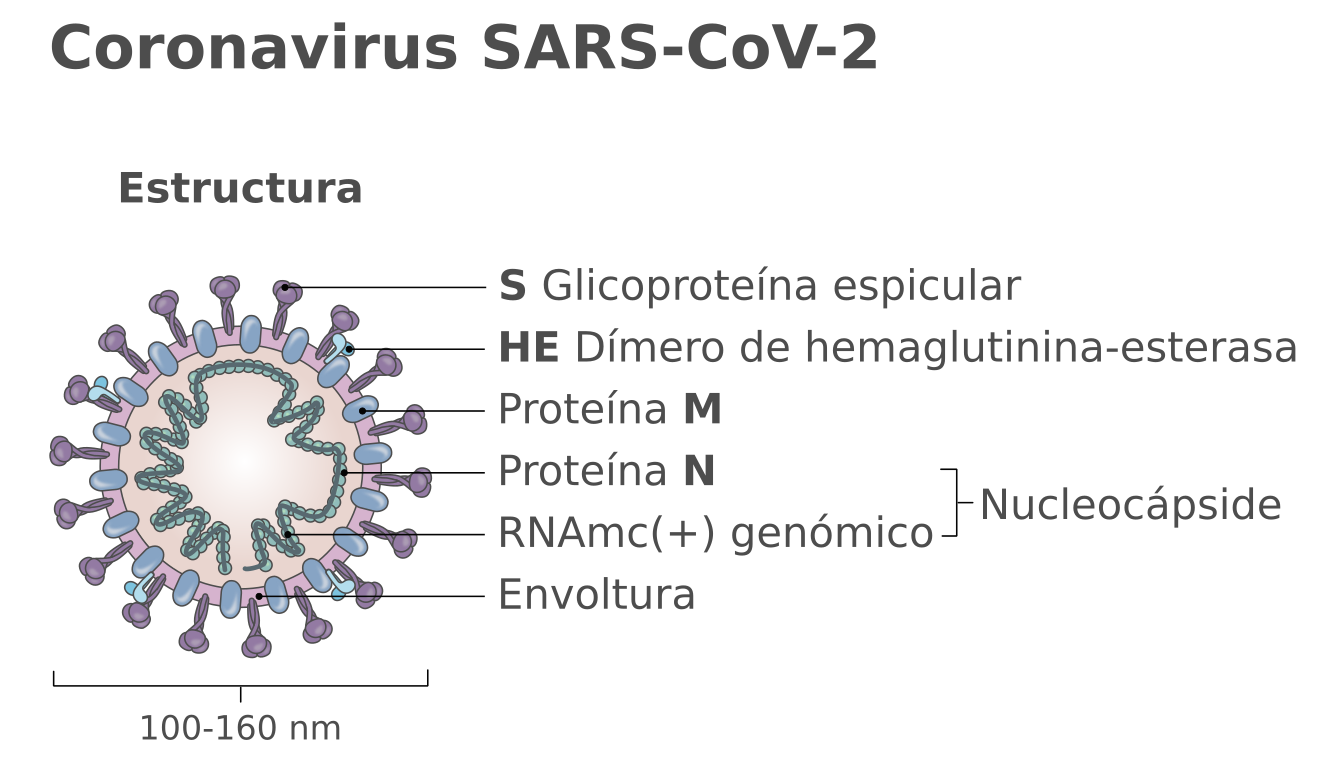
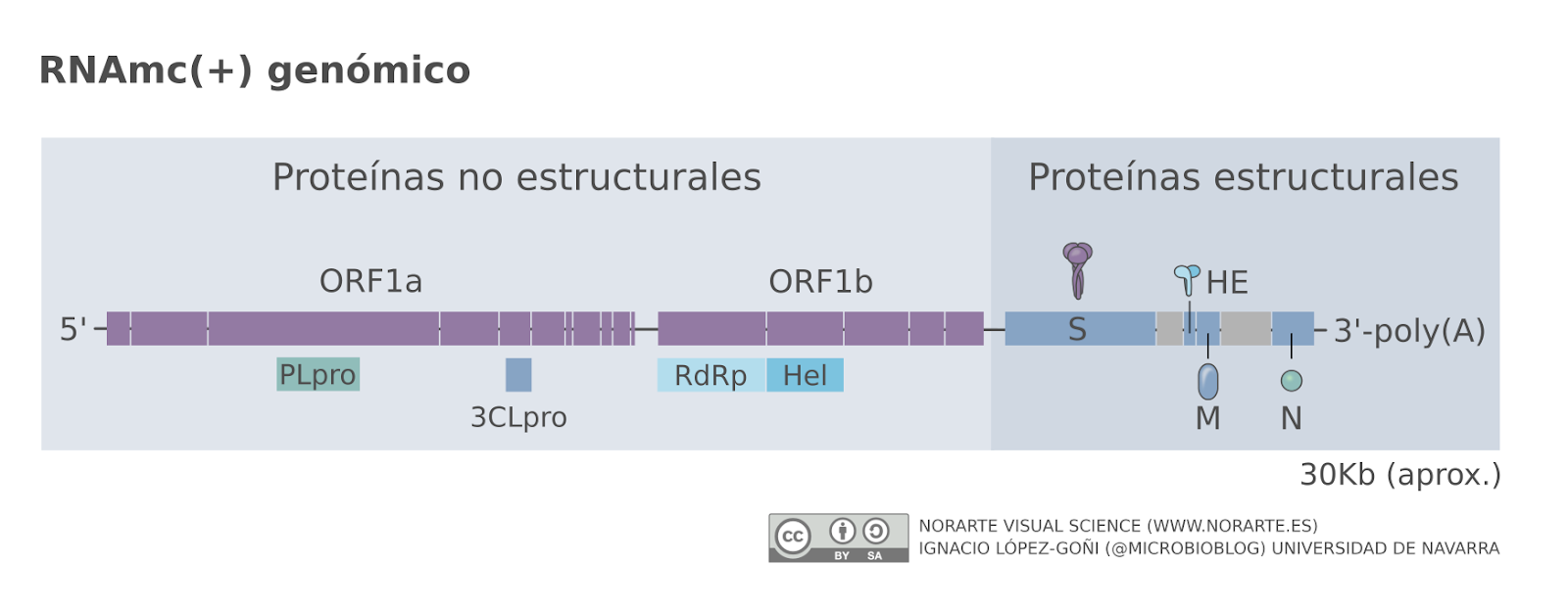
Estructura y genoma del SARS-Cov-2 (Fuente).
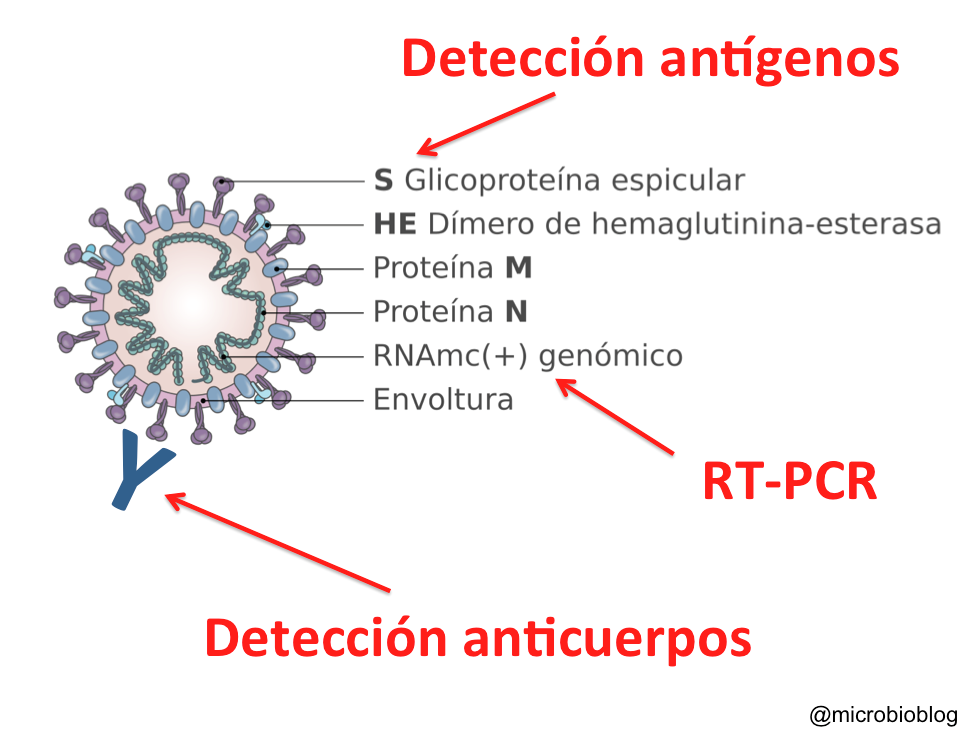
Una de las zonas del genoma más interesantes es la que codifica para la proteína S, porque es la más variables y porque su función es esencial para la entrada en la célula. La proteína S (de spike) forma esas espículas que se proyectan hacia al exterior y que le dan el nombre al corona-virus. El SARS-Cov-2 inicia la entrada en las células humanas después de que la proteína S se una al receptor de la membrana celular, que en este caso es el ACE2. La función biológica de este receptor ACE2 es la maduración de la angiotensina, una hormona que controla la vasoconstricción y la presión arterial. ACE2 es una proteína de membrana que se expresa en pulmones, el corazón, los riñones y el intestino.
La proteína S es la llave de entrada del virus a la célula y la cerradura en la célula es el receptor ACE2. Los modelos en 3D demuestran que en este proceso, la proteína S se divide en dos subunidades, S1 y S2, que se separan por la acción de una enzima de la célula con actividad proteasa, que se denomina furina. Así, S1 se une a su receptor ACE2 y el otro fragmento S2 es escindido a su vez por otra proteasa de la superficie de la célula humana, denominada TMPRSS2. Como resultado la envoltura de virus se fusiona con la membrana de la célula y el virus entra en su interior. Por tanto, la subunidad S1 se encarga de la unión al receptor, mientras que S2 es responsable de la fusión de las membranas.
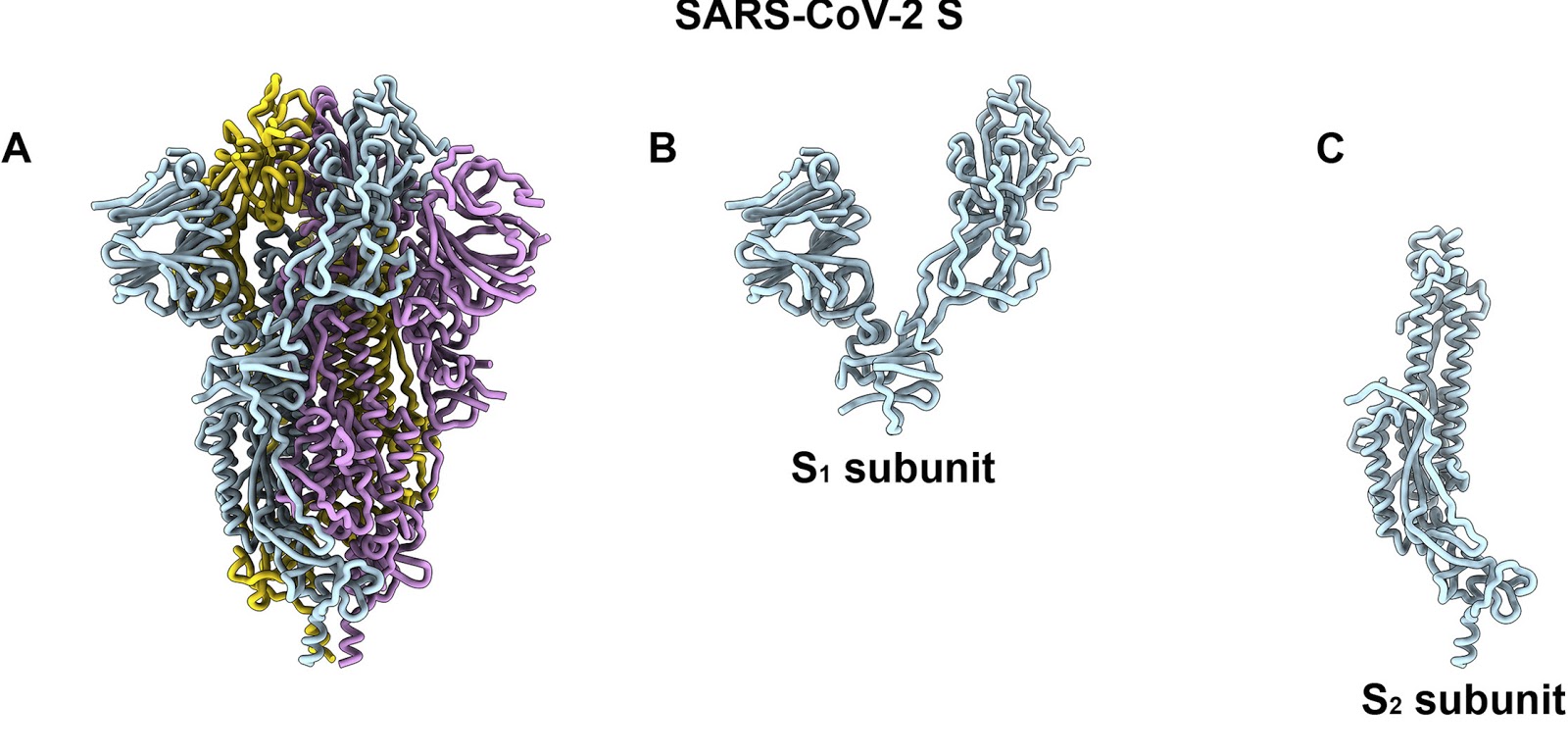
Modelo en 3D de la proteína S de SARS-Cov-2 (Fuente).
Los análisis estructurales, genómicos y bioquímicos de esa proteína S nos permiten estudiar este proceso en detalle y demuestran que SARS-Cov-2 posee dos particularidades importantes.
El dominio RBD de la proteína S tiene una alta afinidad por el receptor ACE2
En primer lugar, la proteína S de SARS-Cov-2 posee una secuencia que se denomina RBD (dominio de unión al receptor), la parte más variable del genoma del virus, en la que hay seis aminoácidos que son esenciales para unirse al receptor ACE2. Si comparamos esa secuencia entre SARS-Cov-2 y el otro coronavirus humano SARS, solo un aminoácidos de esos seis es común. La proteína S de SARS-Cov-2 tiene, por tanto, un dominio RBD que se une con una muy alta afinidad al receptor ACE2 de humanos, pero también de otras especies animales con una alta homología en ese receptor, como hurones o gatos. Esta alta afinidad por el receptor ACE2 probablemente influye es la alta capacidad de infectar las células que tiene este virus. Sin embargo, los análisis computacionales indican que ese dominio no es el mejor posible para unirse al receptor, teóricamente puede haber otras combinaciones que sean aún más eficaces para unirse al receptor. Esto sugiere que esa secuencia ha surgido por un proceso de selección natural a lo largo de pases del virus entre personas o animales. Si fuera un producto manipulado por ingeniería genética, lo habríamos hecho mejor. Si alguien hubiera diseñado este nuevo virus para que fuera patógeno lo hubiera hecho mejor.
La proteína S posee una secuencia de corte por furina
La otra particularidad de la proteína S de SARS-Cov-2 tiene que ver con el sitio de unión entre esas dos subunidades, S1 y S2, de las que está formada. En SARS-Cov-2 esa proteína S tiene una secuencia entre esas subunidades que permite el corte por la enzima de la célula, la furina, y por otras proteasas. Eso determina la infectividad del virus y su rango de hospedador, a qué células o animales puede infectar. Aunque algunos coronavirus humanos, como el HKU1, también tienen esa característica, el sitio de corte por furina no es muy frecuente en todos los coronavirus, y menos en los del grupo beta, al que pertenece el SARS-Cov-2. No sabemos todavía qué consecuencias funcionales puede tener esta propiedad, pero sería importante saber si afecta a su transmisibilidad y patogénesis. Esta secuencia tan peculiar, ¿podría ser fruto de la manipulación genética del virus? Si lo comparamos con lo que ocurre en el virus de la gripe, muy probablemente se haya generado también por selección natural.
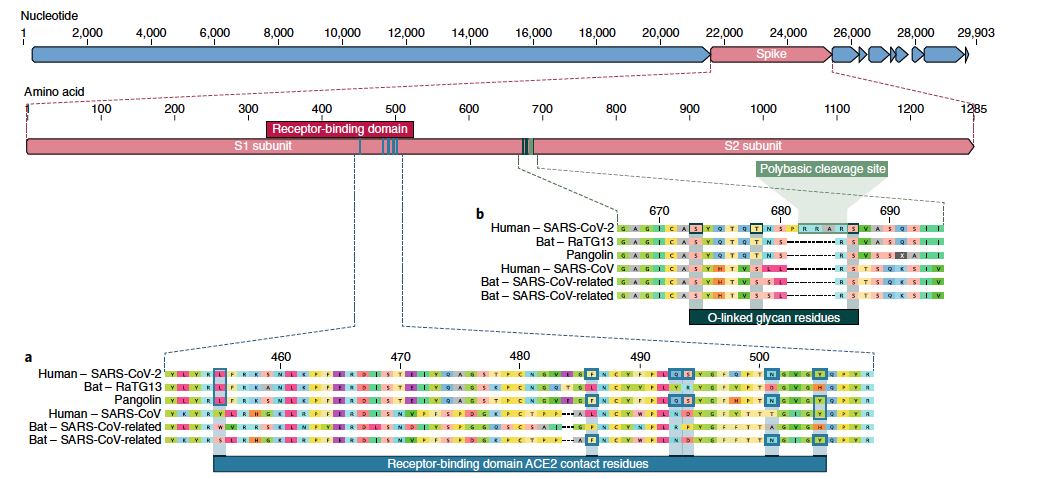
Características de la proteína S de SARS-Cov-2 y otros coronavirus relacionados. Se detalla de forma progresiva la secuencia de nucleótidos del genoma, la secuencia de aminoácidos de la proteína S con sus dos subunidades S1 y S2, el dominio de unión al receptor (RBD) y la zona de corte por furina (polybasic clevage site). (Fuente)
En algunos virus de la gripe aviar se ha visto que en situaciones de alta densidad de poblaciones de aves, se selecciona de forma natural este tipo de secuencias de corte en la hemaglutinina de la envoltura (similar a la proteína S del coronavirus). Esto hace que el virus se replique más rápidamente y sea más transmisible. Así es cómo algunos virus de gripe aviar de baja patogenicidad se convierten en virus de alta patogenicidad. También se ha observado la adquisición de estos sitios de corte en la hemaglutinina después de pases repetidos del virus en cultivo celular o en animales. Por lo tanto, esta nueva propiedad es fruto de la selección natural. Lo mismo ha podido ocurrir en el coronavirus.
Si el origen del genoma de SARS-Cov-2 fuera la ingeniería genética, muy probablemente se habrían empleado algunos sistemas genéticos ya presentes en otros beta-coronavirus y los datos no demuestran nada de esto. Por el contrario, lo más probable es que estas dos características del virus sean fruto de la selección natural y para ello hay dos posibles escenarios: que se haya seleccionado en un animal antes de transferirse al ser humano; o que la selección haya ocurrido en el ser humano después de su transferencia desde un animal.
Selección en una animal antes de transferirse a humanos
Desde el inicio, el origen de SARS-Cov-2 se ha relacionado con el mercado de animales vivos de Wuhan. Cuando se comparan los genomas de los coronavirus, el más parecido al SARS-Cov-2 es el aislado de un murciégalo en Yunnan (China) en 2013, el genoma RaTG13 de Rhinolophus affinis, con más de un 96% de identidad. Sin embargo, cuando se compara la zona RBD de la proteína S difieren significativamente. En otros estudios, se han analizado muestras de varios pangolines (Manis javanica) que llegaron a China por contrabando entre 2017 y 2018, y han detectado coronavirus con una similitud entre el 85 y el 92% con el SARS-Cov-2. Aunque el virus del murciélago sigue teniendo una homología a nivel del genoma mayor, la similitud entre el SARS-Cov-2 y los coronavirus del pangolín era especialmente alta en el dominio RBD de la glicoproteína S, incluidos los seis aminoácidos característicos de esa zona en SARS-Cov-2. Esto refuerza la idea de que la optimización de la proteína S para unirse al receptor ACE2 humano es fruto de la selección natural y no de ingeniería genética o de pases sucesivos del virus en un laboratorio.
Sin embargo, ni los coronavirus de murciélagos ni los de los pangolines tienen el sitio de corte de furina en la proteína S. Los coronavirus son muy frecuentes entre estos y otros animales y es muy probable que todavía no hayamos dado con el precursor animal del SARS-Cov-2. No podemos descartar que fenómenos de mutación, inserción y deleción hayan ocurrido de forma natural en el gen S en algún otro animal, probablemente con alta densidad de población y con un receptor ACE2 similar al humano.
Selección en humanos después de su transferencia desde un animal
Otra posibilidad es que el SARS-Cov-2 haya adquirido esas características mientras se transmitía de forma indetectable entre humanos. Todos los genomas de SARS-Cov-2 secuenciado hasta ahora demuestra que tienen un origen clonal a partir de un ancestro común en Wuhan, muy probablemente a principios de noviembre de 2019. La presencia en los pangolines del mismo dominio RBD en la proteína S sugiere que esa característica ya estaba en el virus antes de su salto a humanos. Quizá, entonces, el sitio de corte por furina fue el que se seleccionó durante la transmisión entre humanos. Esto presupone que el virus estaba presente antes de noviembre de 2019 y que se transmitía entre nosotros de forma indetectable durante un tiempo. Eso ahora no lo sabemos, pero sería muy interesante si somos capaces de hacer estudios retrospectivos y comprobar si realmente el virus circulaba entre nosotros antes de su estallido en Wuhan a finales de 2019.
El hecho de que SARS-Cov-2 entró en los seres humanos a partir de un origen animal implica que la probabilidad de futuros brotes es muy alta, ya que virus similares siguen circulando en la población animal y podrían volver a saltar a los seres humanos.
Conclusión: como vemos las peculiares características de SARS-Cov-2 ya estaban en la naturaleza y no hay que imaginar experimentos de laboratorio para explicar su origen. Conocemos menos del 1% de los virus que hay ahí fuera y más del 70% de los nuevos virus emergentes tienen su origen en los animales. Los virus son millones de millones de “individuos”, que se multiplican a una velocidad enorme y con una frecuencia de mutación y recombinación extraordinaria. Los virus no es que muten, es que viven mutando. En ellos, la evolución va a cámara rápida. La naturaleza tiene suficientes recursos como para generar este y otros muchísimos virus.
Referencias:
– The proximal origin of SARS-CoV-2. Andersen, K.G., et al. Nat Med (2020). https://doi.org/10.1038/s41591-020-0820-9
– Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2.
Renhong Y., et al. Science 27 Mar 2020. Vol. 367, Issue 6485, pp. 1444-1448.







Hola profesor
A cuantos años o decadas estamos de si poder hacer un virus como este en laboratorio?
Segunda pregunta cuando la curva vuelva al punto inicial cero casos nuevos por dia y sin cuarentena, porque no volvera a crecer la curva y sera una repeticion vovler a poner cuarentena asi hasta la vacuna?
Primera: ya tenemos la tecnología para crear virus en el laboratorio.
Segunda: no sabemos si habrá una segunda oleada del virus, pero quizá si hay más gente ya inmunizada la incidencia será menor.
Muchas gracias por todas los interesantes posts que subes a tu blog. En relación a este último y en el supuesto de una selección en humanos después de su transferencia desde un animal, comentas que esto presupone que el virus estaba presente antes de noviembre de 2019 y que se transmitía entre nosotros de forma indetectable durante un tiempo. En este caso ¿piensas que una parte de la población ya podría estar inmunizada? ya que la mutación en el sitio de corte de la furina supone un fragmento pequeño en el conjunto de la estructura de la proteína S y del resto de proteínas de superficie del virus. Una vez más muchas gracias!
Hasta que no se haga un estudio serológico, detección de anticuerpos, en la población no los sabremos. Gracias a ti!
Saludos desde Ecuador Dr. Le tengo una petición, ¿ podría usted hablar sobre zoonosis y sus distintos tipos para entender mejor el origen del SARSCOV2?
Un post muy interante sobre el SASR-CoV-2. Aquí dejo una duda:
– Los SARS, y este virus en particular, presentan HE en su superficie? Creo que sólo se presentan en los del tipo A, HCoV-OC43 y HCoV-HKU1.
Gracias y saludos!
Hola, gran pregunta. Pero quisiera que alguien me responda que saben sobre los fragmentos Hr1 y Hr2 del virus. Gracias.
Excelente explicación… Muchas gracias
Hola, por que presenta tanta dificultad crear un inhibidor enzimatico especifico para la proteasa del virus?
Habría alguna forma de saber si un genoma de virus ha sido editado?
O es imposible saber diferenciar entre la edición y las mutaciones que ya tiene de por sí el virus?
Muchas gracias por los artículos.
Me encantó la charla de Naukas Bilbao.
Gracias por la docencia.
Por supuesto que el virus puede tener un origen natural, pero también puede ser artificial. Precisamente en el artículo de Andersen et al. en el epigrafe "3. Selection during passage" se expone que no hay trabajos descritos en la literatura sobre una mejora en laboratorio como la zona de corte por furina en este tipo de virus. ¿En serio se piensa que un laboratorio militar chino/estadounidense publicaría sus resultados en este campo?
El coronavirus proviene de alguna criatura de las cuevas??.?Mi hijo mas o menos en la época de la H1n1 contrajo primero H1n1 y a los meses visitamos la cueva de las lechuzas y volvimos e hizo una infección similar al del Coronavirus sano de milagro dicen por los anticuerpos que desarrolló de la H1n1 pero se de casos de otras gentes que visitaron esas cuevas que murieron de neumonia o algo así.
Leerles me ilustra. Gracias por su dedicación. No tengo ni idea del tema, soy ingeniero mecánico. Muy amigable el artículo. Saludos Roberto Alarcón desde Quito.
En la década de 1920, había unos 10.000 millones de conejos silvestres en Australia, según los cálculos del Gobierno.
Las autoridades han propagado ahora, en unos 600 puntos de la isla, la nueva cepa K5 del conocido virus de la enfermedad hemorrágica del conejo. La cepa, denominada científicamente RHDV1 K5, se ha aislado en Corea del Sur y solo afecta a los conejos silvestres. “Es mucho peor que el ébola. El virus resiste meses activo en el medio ambiente. Los conejos mueren en 48 horas y la mortalidad alcanza el 90%”, explica el virólogo Francisco Parra.UNIVERSIDAD DE OVIEDO
https://elpais.com/elpais/2017/05/24/ciencia/1495644688_435337.html
Felicidades por su blog, es ameno e instructivo.
Desde que se están publicando datos sobre cómo el virus mantiene su capacidad de infección durante horas, e incluso días, permaneciendo sobre objetos inertes me surge la pregunta de ¿cómo es posible?. Solo está rodeado de una membrana plasmática, no fabrica ninguna forma de resistencia ante la sequedad o el oxígeno y sin embrago mantiene su estructura, según parece. ¿Podría explicar el por qué?
Muchas gracias.
Buenas tardes, me ha aliviado leerle, pero me quedan algunas dudas. Puedo escribirle? juancarlos.lopez1@correo.gob.es
Hola Profe, ha leido este articulo "A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence", publicado en la revista Nature Medicine en noviembre del 2015, aparece la Dra Zhengli-li Shi, ex-directora del laboratorio wuhan Nivel 4 y una autoridad mundial en Coronavirus.
Los modelos en 3D demuestran que en este proceso, la proteína S se divide en dos subunidades, S1 y S2, que se separan por la acción de una enzima de la célula con actividad proteasa, que se denomina furina.
Así, S1 se une a su receptor ACE2 y el otro fragmento S2 es escindido a su vez por otra proteasa de la superficie de la célula humana, denominada TMPRSS2
ES LA MISMA PROTEASA LA FURINA QUE LA TMPRSS2 SOLO ESO. GRACIAS.
Gracias por el comentarios, pero yo no lo tengo tan claro, ¿puedes adjuntar alguna referencia, por favor?
Agradezco la información
En su primera imagen hay un error, esa no es representación 3D de la Proteína S, es de la proteína ACE-2
Hola, un par de preguntas: La secuencia de aminoacidos de la proteina S es la misma para todas las posibles cepas que haya del virus ?, es decir, las vacunas de ARN mensajero valdrian para todas las cepas que pudiera haber del virus ?