El control de las enfermedades infecciosas causadas por bacterias patógenas
depende de un diagnóstico rápido y
preciso. Según el tipo de microorganismo, es necesario su cultivo en el
laboratorio lo que puede resultar peligroso y en muchos casos necesitar varios
días e incluso semanas. Algunos microorganismos simplemente no crecen en los
medios de cultivo. Desde hace ya muchos años se han ido desarrollando ensayos moleculares
basadas en la amplificación específica del genoma de la bacteria mediante la
técnica de la PCR (Polymerase Chain Reaction,
pincha aquí si quieres ver un vídeo [1:47] que explica cómo funciona la
PCR).
Son decenas
de miles los artículos científicos publicados sobre el empleo de esta técnica
para detectar patógenos microbianos. Pero la reacción de PCR también tiene
algunas limitaciones: emplea componentes enzimáticos muy sensibles que
requieren una cuidadosa purificación de los ácidos nucleicos (ADN o ARN) de la
bacteria, y sofisticados y caros termocicladores. Además, la técnica requiere ciertas
habilidades por parte del personal. Por eso, sigue siendo un reto el
desarrollar tecnologías sencillas y baratas que permitan un diagnóstico rápido
y preciso.
Un
grupo de investigadores canadienses han desarrollado un pequeño dispositivo que
combina un sistema para romper y lisar las bacterias con un biosensor
electroquímico ultrasensible capaz de detectar la bacteria.
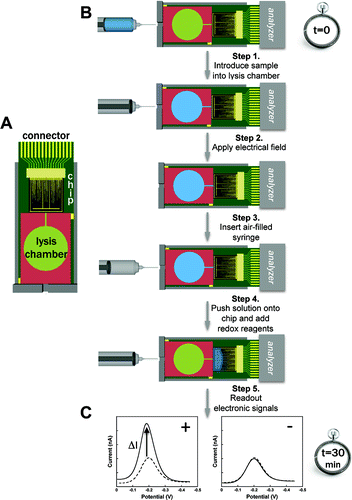
El chip
incluye un biosensor que contiene unas sondas (monocadenas de ADN específicas)
que al unirse o hibridarse con el ADN complementario de la bacteria generan una
respuesta electrocatalítica que se traduce una señal eléctrica detectable.
El
sistema integra en un único dispositivo todo lo necesario. Primero se introduce
a través de una jeringa en una cámara de
lisis la muestra problema, que contiene las bacterias. Se aplica una descarga eléctrica capaz de
romper y lisar las bacterias, lo que libera sus ácidos nucleicos. La muestra
pasa al chip de detección que
consiste en unos microelectródos que detectan secuencias de ácidos nucleicos
específicas que se unen a sondas moleculares inmovilizadas en el sensor. Esta
reacción de hibridación genera una señal eléctrica fácilmente detectable en el
sensor. El proceso se completa en menos
de 30 minutos.
El chip
lo han validado con muestras de Escherichia coli (una bacteria
Gram-negativa) y de Staphylococcus (Gram-positiva y más resistente a la lisis).
Comprobaron que la descarga eléctrica era capaz de romper ambas bacterias y
liberar sus ácidos nucleicos. Y mediante hibridación con sondas específicas
detectaron cantidades tan pequeñas como 1 bacteria por microlitro! También lo
ensayaron con muestras de orina contaminadas con las mismas bacterias.
Es el
primer sistema de detección “PCR-free” basado en un chip que incluye una cámara
de lisis acoplada a un microsensor ultrasensible. Todos los componentes del sistema son muy
baratos y están integrados en un solo dispositivo muy fácil de usar.
Aunque
es necesario realizar más pruebas con diferentes tipos de microorganismos, a
distintas concentraciones y en muestras biológicas reales, no cabe duda que
este nuevo chip “prodigioso” puede ser
el comienzo de una nueva revolución en la detección de microbios patógenos,
como lo fue en su día la PCR: la “vieja” PCR que quizá ahora pase ya a los
libros de historia!
Si te
ha gustado esta entrada quizá te interese esta otra también sobre microchips: Un sueño hecho realidad: diagnosticar enfermedades infecciosas en mitad de la sabana africana por unos céntimos de Euro en unos pocos minutos.
Lam, B., et al. (2012). Polymerase Chain Reaction-Free, Sample-to-Answer Bacterial Detection in 30 Minutes with Integrated Cell Lysis Analytical Chemistry, 84 (1), 21-25 DOI: 10.1021/ac202599b








Y publicar los resultados por Twitter …
y desde un IPhone… sigo siendo de los microbiologos mañosos de vieja escuela jojojojoj
Buen post me gustó mucho. Gracias.
Gracias a ti, por seguir.
Espero que pase la etapa de prototipo.
Ojalá!
Buen día. Podríamos tener un falso positivo, por ejemplo en perros que hayan sido recientemente vacunados, con un biológico que use como antígeno una bacterina (con la bacteria completa inactivada) de varios serovares de leptospira??
Gracias por tu interés, pero lamentablemente no te sé responder. Lo siento.