En 1928 Alexander Fleming observó que un hongo (Penicillium) que creció accidentalmente sobre unas placas de
cultivo con la bacteria Staphylococcus,
creaba un círculo libre de bacterias alrededor suya. Descubrió así una
sustancia producida por el hongo capaz de inhibir el crecimiento bacteriano, a
la que denominó penicilina, el primer antibiótico.
Los microbios son capaces de producir
y secretar compuestos químicos naturales con múltiples efectos sobre otros
seres vivos. Gran parte de la industria farmacéutica se ha basado sobre eldesarrollo de estas pequeñas moléculas
naturales producidas por bacterias y hongos. Descubrir nuevas moléculas con
actividad interesante es un trabajo inmenso que requiere mucho tiempo de
dedicación e inversión. Desde el punto de vista económico a veces no es
rentable y esto hace que algunas empresas farmacéuticas hayan dejado de
investigar y producir nuevos antibióticos, por ejemplo.
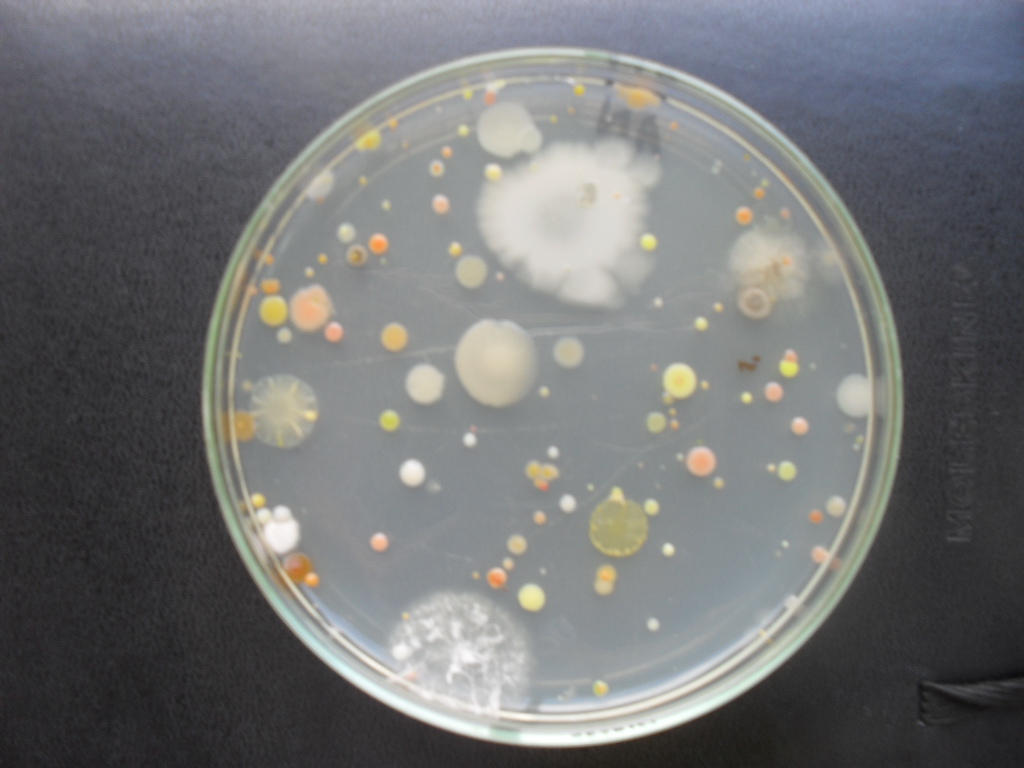
Las bacterias y muchos hongos crecen
sobre la superficie de agar
de los medios de cultivo formando agrupaciones de microorganismos
o colonias (Fuente).
Un grupo de investigadores han desarrollado
una novedosa tecnología rápida y de alta sensibilidad que puede revolucionar la manera de buscar nuevos
productos químicos de origen microbiano. Para ello, analizan directamente sobre
las colonias bacterianas las moléculas secretadas mediante técnicas de espectroscopia de masas, pudiendo así
identificarlas y caracterizarlas.
La técnica se basa en un sistema de
espectroscopia de masas denominad nanoDESI (nanospray
desorption electrospray ionization) que se
combina con la construcción de redes moleculares para la identificación de los
compuestos. Mediante un capilar se añade un solvente directamente sobre cualquier
superficie de estudio. Otro capilar recoge las moléculas liberadas que son
enviadas al espectrómetro de masas para su análisis.
Han aplicado esta técnica sobre la
superficie de colonias de bacterias vivas creciendo sobre una placa de Petri,
sin necesidad de manipular o preparar la muestra. De esta forma han analizado
la producción de compuestos químicos a partir de colonias bacterianas de
distintos tipos, como Bacillus subtilis,
Streptomyces coelicolor, Mycobacterium smegmatis y Pseudomonas aeruginosa. Han encontrado una
increíble diversidad de moléculas, que sobrepasa por mucho nuestra actual
visión del metabolismo bacteriano secundario.
Por ejemplo, mediante el análisis de
los compuestos producidos por una sola colonia de Bacillus subtilis a distintos tiempos, han sido capaces de observar
la activación y supresión temporal de compuestos concretos del metabolismo de
la bacteria. Aunque muchas de las moléculas detectadas ya se conocían, otras
son completamente nuevas o son variantes estructurales de compuestos ya
conocidos. La cantidad de compuestos
químico que una bacteria es capaz de producir es muchísimo mayor de los que se
creía, mucho más compleja y más sofisticada, lo que demuestra la enorme
plasticidad biosintética de las bacterias.
Se han secuenciado ya muchos genomas
bacterianos, pero no conocemos la función de la mayoría de sus genes. Mediante
esta técnica se podrían relacionar determinados genes con funciones metabólicas
concretas. Además, permitirá descubrir de manera mucho más rápida y barata
nuevos compuestos químicos de origen microbiano que podrán revolucionar la
industria farmacéutica, como en su día ocurrió gracias a la observación casual
de Fleming.
Mass spectral molecular networking of living microbial
colonies. Watrous J, et
al. ProcNatlAcadSci USA. 2012.
109(26):E1743-52.