No podemos descartar que en las próxima semanas o meses vayan apareciendo nuevas variantes
En las últimas semanas se está hablando mucho de las nuevas variantes del SARS-CoV-2. ¿Qué sabemos hasta ahora?
Mejor hablamos de variantes genética, no de cepas. Variante implica diferencias en la secuencia del genoma, debido a mutaciones. Cepa es una variante en la que se demuestran cambios importantes en su biología (antigenicidad, transmisibilidad, virulencia, …).
Los virus mutan constantemente, viven mutando. Una población de virus es una nube de mutantes, con pequeñas diferencias genéticas. Se han detectado ya varios miles de mutantes de SARS-CoV-2, la mayoría sin ningún efecto.
Se ha calculado que la frecuencia de mutación del SARS-CoV-2 es de dos mutaciones al mes aproximadamente, esto supone que las variantes que ahora circulan pueden haber acumulado unas 22 mutaciones, respecto a la secuencia original del primer aislamiento de Wuhan.
De todas las mutaciones las que más preocupan son las que afectan al gen que codifica para la proteína S, porque es la que interacciona con el receptor celular ACE2 (la puerta de entrada a la célula). Además, al ser la proteína más expuesta es también la más antigénica, sobre la que actúan los anticuerpos. La mayoría de las vacunas utilizan esta proteína como su estrategia para activar el sistema inmune.
En las últimas semanas se han detectado nuevas variantes con distintas mutaciones en el gen S. Probablemente la presión a la que estamos sometiendo al virus (personas que ya tienen anticuerpos porque han pasado la infección, medidas para evitar la transmisión, las vacunas, …) esté forzando a que se seleccionen mutantes más transmisible. O quizá simplemente sea un fenómeno al azar.
Las tres variantes que más preocupan en este momento son:
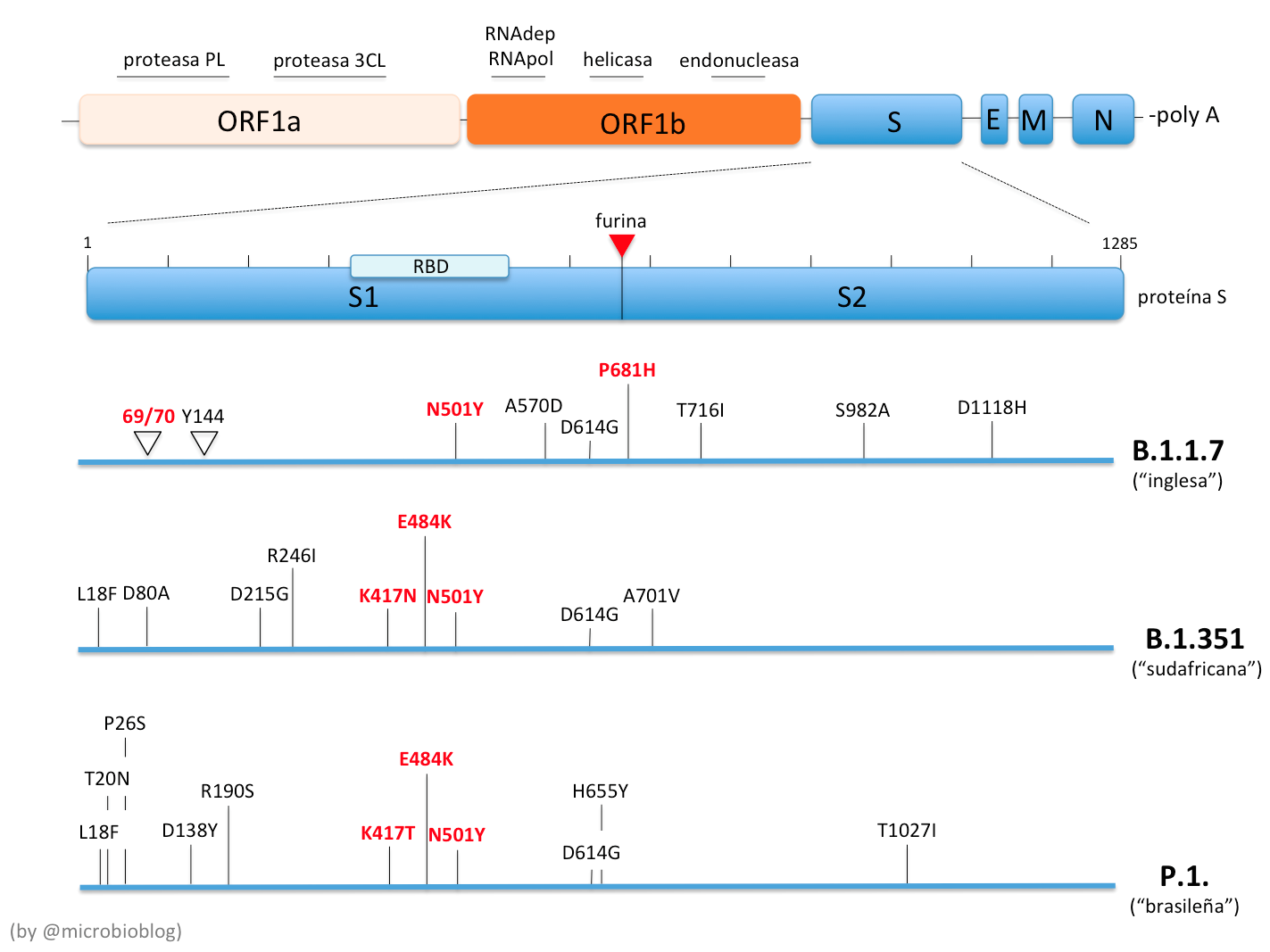
Las mutaciones en la proteína S de las tres variantes B.1.1.7, B.1.351 y P.1. En la parte superior se muestra un esquema de la organización del genoma de SARS-CoV-2. Debajo se detalla la proteína S, con sus dos dominios S1 y S2, la región especifica de unión al receptor (RBD, Receptor Binding Domain, en el dominio S1) y la zona de corte por furina. Las mutaciones 69/70 y Y144 son deleciones en la variante B.1.1.7. En rojo se señalan las mutaciones más importantes. Algunos le han puesto apodo a las mutaciones: Nelly a la mutación N501Y y Erik a la E484K. Los números hacen referencia al número del aminoácido en la proteína y las siglas al tipo de aminoácido. Así, por ejemplo, N501Y significa que en la posición 501 se ha sustituido el aminoácido Asparragina (N) por la Tirosina (Y). Otras letras: A, alanina; R, Arginina; D, ácido aspártico; C, cisteína; Q, glutamina; E, ácido glutámico; G, glicina; L, leucina; K, lisina; M, metionina; F, fenilalanina; P, prolina; H, histidina; T, treonina; I, isoleucina; S, serina; W, triptófano; V, valina. (Figura elaboración propia)
B.1.1.7, denominada variante “inglesa”, porque se detectó por primera vez en el Reino Unido en septiembre de 2020 (otras denominaciones VOC 202012/01, 201/501Y.V1). Desde entonces se ha detectado en 62 países (OJO, la extensión depende mucho de la capacidad de secuenciación de cada país: si no se busca no se encuentra). Tiene unas 17 mutaciones, de las cuales 9 están en el gen S. Las que más preocupan son la mutación N501Y que afecta a la región de unión al receptor (RBD), la deleción 69/90 porque causa un cambio en la conformación de la proteína y la P681H porque está cerca de la zona de corte S1/S2 por furina. Se ha sugerido que esta variante es más transmisible (se transmite de forma más eficiente y más rápido) y que hay una probabilidad real de que sea más letal. No parece que afecte de momento a la reactividad con anticuerpos ni a las vacunas actuales. NOTA (4/2/2021): ya se han detectado en Reino Unido algunos aislamientos de esta variante que han adquirido la mutación E484K (ver más adelante).
B.1.351, denominada variante “sudafricana”, porque se detectó por primera vez en Sudáfrica en octubre del 2020 (otras denominaciones 20C/501Y.V2). Se ha detectado en al menos 26 países. Tiene unas 21 mutaciones, de las cuales 9 están en el gen S. Comparte algunas mutaciones (como la N501Y) con la variante “inglesa”, pero preocupa porque además tiene otras mutaciones en la misma región RBD: E484K y K417N. La mutación E484K supone un cambio de aminoácido asociado a un cambio de carga (un aminoácido con carga negativa se sustituye por otro con carga positiva). Esto, junto con la mutación N501Y, puede afectar a la unión del virus a la célula. De momento, no hay datos sobre su transmisibilidad y no sabemos si es más virulenta, pero preocupan algunos datos preliminares que sugieren que podría escapar de la neutralización por anticuerpos tras una infección natural o la vacunación.
P.1., denominada variante “brasileña”, porque se detectó a principios de año en Japón en cuatro viajeros procedentes de Brasil (otras denominaciones 20J/501Y.V3, B.1.1.28.1). Tiene unas 17 mutaciones, de las cuales 10 están en el gen S. Se ha detectado en al menos 7 países. Presenta también las mutaciones N501Y y E484K y otra similar K417T. No hay datos, de momento, sobre su transmisibilidad, virulencia o reacción con anticuerpos. Recientemente se ha descrito en la región de Manaus (Brasil) una alta incidencia de reinfecciones. En esa región hasta un 76% de la población había sido infectada por el virus en la primera oleada, y ahora se han detectado algunas reinfecciones por esta variante. Sin embargo no se puede descartar que las reinfecciones no sean debidas a las propiedades de la variante, si no a otras circunstancias, como la falta de medidas para evitar el contagio.
La aparición de nuevas variantes no debe sorprendernos, es evolución en estado puro. Conforme le “metemos” más presión al virus, este sigue evolucionando y se van seleccionando aquellas variantes que escapan de ese presión. Es por tanto muy probable que en las próxima semanas o meses vayan apareciendo incluso nuevas variantes. De hecho ya se han descrito algunas nuevas: COH.20G/501Y y S Q677H, en EE.UU.; L452R en EE.UU. y en Europa. Por eso, denominarlas como variantes «inglesa», «sudafricana» o «brasileña» no tienen mucho sentido. La detección de las variantes depende de la capacidad de secuenciación de cada país. Irán apareciendo nuevas y es fundamental incrementar la vigilancia y la secuenciación de los aislamientos, para identificar qué variantes están circulando en cada país y poder hacer un seguimiento de los nuevos mutantes. Dudo de que las medidas como el cierre de fronteras con países concretos (o la cancelación de vuelos internacionales) tenga sentido y evite la extensión de estas variantes, que pueden surgir en cualquier sitio. Es necesario investigar qué efecto pueden tener estas variantes en la virulencia del virus y si se relaciona con una mayor gravedad de la enfermedad, o con un mayor número de reinfecciones.
Es pronto todavía para saber cómo estas variantes podrían influir en la efectividad de las vacunas. Hay que tener en cuenta que la mayoría de las vacunas inducen anticuerpos neutralizante contra varias zonas de la proteína S, además de activar la inmunidad celular (que en el caso de lo virus es esencial), así que es improbable que una mutación puntual pueda cambiar la efectividad de las vacunas. Sin embargo, la acumulación de mutaciones en zonas criticas de la proteína S, como la RBD u otras que pueden cambiar la conformación de la proteína, sí que pueden disminuir la eficacia de las vacunas. Es algo que habrá que seguir evaluando muy de cerca.
Por último, todo esto refuerza la idea de la importancia de las medidas de contención del virus. En este sentido, retrasar las vacunaciones (por falta de suministro o por una gestión deficiente) no es una buena noticia: cuanto antes vacunemos a la población mucho mejor, tener grupos sin vacunar o mal vacunados podría favorecer la aparición de nuevas variantes. Además de la «presión selectiva» también hay tener en cuenta el número. A una misma tasa de mutación, cuantos más viriones haya, más mutantes habrá. Por lo tanto, cuántos más infectados haya, muchos más millones de partículas virales habrá, y muchos más mutantes se generarán. Si el mutante aparece en un individuo que no infecta, desaparece. Pero si aparece en uno que infecta, se expande. Más infectados, más mutantes. Esta es otra razón para que vacunemos y disminuya el número de infectados: a mayor número de infectados más probabilidades hay de que aparezcan variantes mutantes severas. Mantengamos las normas y evitemos infectar a otros.
Te recomiendo un par de páginas espectaculares con toda la información detallada sobre el genoma, las mutaciones y la variantes genéticas de SARS-CoV-2:
– SARS-CoV-2 Proteome-3D Analysis (University of Cambridge)
– CoVariants (Institute of Social and Preventive Medicine, University of Bern)
NOTA: me comentan varios veterinarios que éste es el problema de las vacunas contra coronavirus en animales. Aunque digamos que los coronavirus mutan «menos» que los virus de la gripe o el VIH, eso no quiere decir que no muten. Por eso, la revacunación en el mundo animal es frecuente.
También te puede interesar:
– La nueva variante “inglesa” de SARS-CoV-2
– La complejidad de las nuevas variantes del coronavirus








A nivel genético, aparte de las polimerasas, que afectaría a la mutabilidad de un virión? Igual es una pregunta muy estúpida pero estoy bastante oxidado en molecular a estas alturas.
En el paper de la cepa B.1.1.7. no había un aumento significativo de mutabilidad, ¿Sigue siendo así?
Felicitaciones Dr. Lopez-Goñi por el informe, siempre leo sus publicaciones que actualiza…un gran saludos desde Salta Argentina…
Excelente artículo e información… gracias por compartir