Los microorganismos no cultivables
aparecen en la nueva versión del árbol de la vida
El árbol de la vida (The tree of life) es uno de los
sistemas de organización de los seres vivos más importantes en biología.
Los primeros intentos de clasificar los organismos en un “árbol de la vida” se
basaba en sus características físicas y metabólicas. Con los métodos
moleculares basados en la comparación de secuencias de genes aumentaron la
diversidad de las ramas del árbol porque ya no era necesario la observación
directa de los organismos. Hasta ahora estas comparaciones se limitaban a un
gen, el de la subunidad pequeña del RNA
ribosomal (SSU rRNA, small subunit
ribosomal RNA). Para ello, a partir de una muestra de DNA del organismo, se
amplificaba el gen SSU rRNA con oligonucleótidos (primers) específicos y universales (en principio para todos los organismos),
se secuenciaba y se comparaba con las secuencias del mismo gen del resto de
organismos. La comparación de las secuencias del gen SSU rRNA demuestra que la
vida se organiza en tres líneas evolutivas, denominadas dominios: Bacteria
y Archaea (que representan células procariotas, es
decir sin núcleo), y Eukarya (células eucariotas, con
núcleo). Este árbol filogenético
universal reveló dos hechos evolutivos importantes: no todos los
procariotas están estrechamente relacionados desde el punto de vista evolutivo,
y el dominio Archaea presenta una relación más próxima al
dominio Eukarya que al dominio Bacteria.
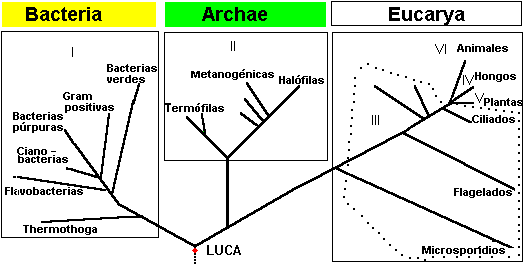
El árbol filogenético universal, basado en la comparación de las secuencias del gen SSU rRNA.
Sin
embargo, este método también tienen sus limitaciones. Por una parte, existen
muchos organismos en la naturaleza (sobre todo microbios) que todavía no somos capaces de cultivar en
el laboratorio y no los podemos aislar y de los que hasta ahora no teníamos
información sobre ellos (la “materia oscura” del mundo microbiano). Por otra, los
primers no son tan universales como
se creía al principio: existen organismos
cuyas secuencias divergen de esos primers
universales y no podemos tener información sobre su gen SSU rRNA.
Existen más de 30.500 genomas
secuenciados de los tres dominios de la vida, Bacteria, Arquea y Eukarya (datos
de septiembre de 2015).
La metagenómica se basa en la secuenciación
masiva de todo el DNA de una muestra ambiental. El resultado son cientos de
miles de secuencias de fragmentos del genoma. Luego, nuevos métodos bioinformáticos
permiten enlazar esos fragmentos (como un puzle) y completar (o casi completar)
la secuencia de todo el genoma de un organismo concreto. Con esta técnica no es
necesario aislar el organismo, no tenemos necesidad de cultivarlo en el
laboratorio. Ni siquiera es necesario tener un genoma de referencia previo para
compararlo. Además, esta aproximación genómica nos proporciona información
sobre el potencial metabolismo del
organismo, información que puede ser empleada para relacionarlo con el
resto de organismos y clasificarlo.
Ahora,
un grupo de californianos (junto con algún japonés) han empleado la tecnología
metagenómica para proponer una nueva versión del árbol de la vida en alta definición,
como si viéramos el árbol con una mayor
resolución. Y el resultado es muy interesante.
Han
construido su árbol de la vida usando unos 2.000 genomas completos obtenidos de
bases de datos públicas más otros 1.011
nuevos genomas reconstruidos a partir de muestras de DNA obtenidas de
diferentes ambientes. Estos eran por tanto genomas de organismos no cultivados en el laboratorio. Las muestras de DNA
las han obtenido de varios ecosistemas: un sistema acuífero superficial, simas
profundas marinas del Japón, cortezas salinas del desierto de Atacama, suelo de
verdes praderas californianas, un geiser rico en CO2 y hasta de la
boca de un par de delfines (como ves los autores se han divertido de lo lindo en
la fase de recogida de muestras).
Una
vez obtenidas las secuencias de DNA y montados los genomas, compararon las de 16 proteínas ribosomales de cada organismo.
De esta forma, obtuvieron un árbol en HD (alta definición), con mucha mayor resolución
que los árboles clásicos obtenido al comparar una sola secuencia del gen 16S rRNA.
Usaron secuencias de proteínas ribosomales para evitar artefactos que se
podrían originar si se emplean genes con funciones distintas y sujetos a
diferentes procesos evolutivos. Además, los genes ribosomales siempre están
localizados juntos en una pequeña región del genoma en Bacteria y Archaea. Para
la construcción del árbol se incluyeron un representante por género de todos
los géneros para los que existe un genoma completo secuenciado (o al menos un
borrador de alta calidad).
En esta nueva versión del árbol
de la vida se han incluido 3.083 organismos.
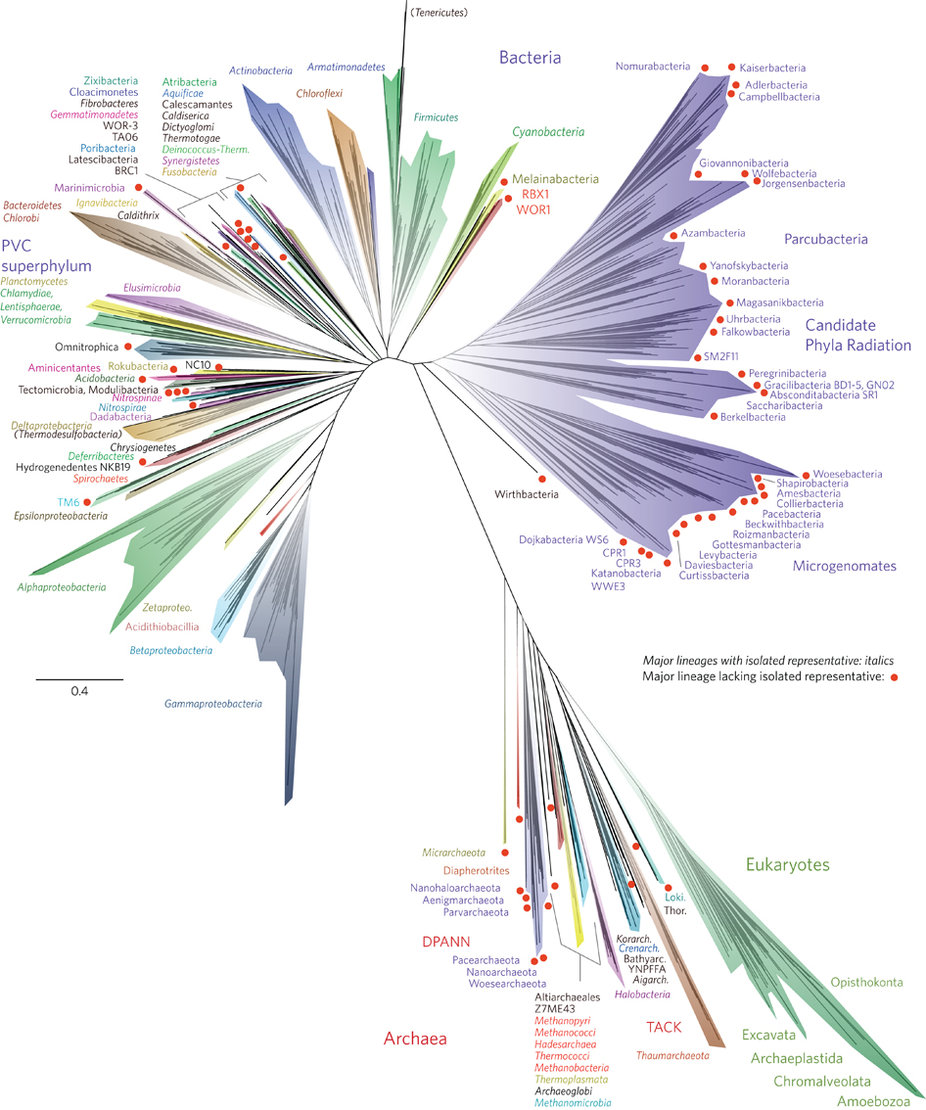
El
nuevo árbol de la vida en alta definición. Incluye 92 phyla de Bacteria, 26 de Archaea, y los cincos supergrupos de Eukarya. Se señala con un punto rojo los
linajes que no tienen un representante aislado y cultivado.
Se trata del primer árbol de la
vida publicado desde el desarrollo de las técnicas metagenómica. Ha requerido
un total de 3.840 horas de trabajo computacional del superordenador CIPRES.
En nuevo
árbol demuestra que el dominio Bacteria
es el que más linajes tiene, el más diverso. La mayor biodiversidad genética se encuentra entre las bacterias. Archaea es menos abundante y menos diverso
que Bacteria. La baja diversidad
genética de Eukarya es esperable, debido a su comparativamente reciente
evolución.
El
resultado es compatible además con la idea de que los eucariotas evolucionaron
como quimeras vía fusiones endosimbiónticas en las que participaron tanto bacterias
como arqueas. El dominio Eukarya
incluye protistas, hongos, plantas y animales, y se ramifica a partir de Archaea, en concreto del grupo TACK. Estos análisis
filogenéticos apoyan la hipótesis de que la arquea Lokiarchaeota y Eukarya
poseen un mismo ancestro común.
Otro
dato interesante, es que a diferencia de los que se pensaba, la clase Proteobacteria del dominio Bacteria, no es un grupo monofilético,
sino que tiene orígenes evolutivos más diversos.
Pero
lo más innovador de este árbol en alta
definición es la aparición en escena de un gran número de linajes sin representante
aislado, no cultivados (los puntos rojos en la figura de arriba). La
mayoría de estos se agrupan además dentro de una misma región del árbol,
denominada CPR (Candidate Phyla Radiation).
Según esto el domino Bacteria se divide claramente en dos
linajes. Los géneros incluidos en este nuevo grupo CPR tiene algunas
características comunes (además de ser no cultivables): todos tiene el genoma
pequeño, la mayoría poseen capacidades metabólicas restringidas, carecen del
ciclo del ácido cítrico, cadena respiratoria y tienen una limitación de
síntesis de nucleótidos y aminoácidos, por lo que muchos son simbiontes. No
está claro si esto es debido a pérdida progresiva de capacidades o que por el
contrario son características heredadas de una forma ancestral de vida con un
metabolismo primitivo muy simple.
En resumen,
la inclusión de nuevos genomas de linajes microbianos previamente desconocidos ha
expandido enormemente el árbol de la vida. Esto demuestra la importancia de
incluir datos genómicos independientes del cultivo para tener una imagen más
real del árbol de la vida.
También te puede interesar:
– El árbol que plantó Carl R. Woese
– Dos mejor que tres: un poda en el árbol de la vida
– Sobre el origen de las células eucariotas
A new view of the tree of life. 2016. Hug, L. A. y col. Nature Microbiol. Article number: 16048 (2016). doi:10.1038/nmicrobiol.2016.48








Excelente contenido! Gracias por la información