Sobrevivir a una infección viral es fundamental para cualquier forma de vida. Para controlar una infección es necesario que el virus invasivo sea capturado por las células para su reconocimiento específico y posterior destrucción. Es lo que se denomina inmunidad adaptativa. En los mamíferos, este sistema se basa en la respuesta antígeno-anticuerpo. Pero las bacterias y las arqueas (los procariotas) no tiene este sistema de antígenos-anticuerpos. ¿Cómo se protegen entonces frente
a una infección viral?
En los procariotas existe también un sistema de defensa que confiere inmunidad adaptativa frente a los virus (bacteriófagos) que les infectan, el denominado CRISPR/Cas del inglés “Clustered Regularly Interspaced Short Palindromic Repeats”, algo así como repeticiones palidrómicas cortas interespaciadas agrupadas regularmente, un nombre que seguro no te dice nada.
CRISPR/Cas es el sistema de defensa inmune adaptativo de las bacterias,
capaz de reconocer un DNA extraño y degradarlo
¿Cómo es el sistema CRISPR/Cas?
El sistema CRISPR/Cas tiene dos componentes: un grupo de secuencias de DNA repetidas varias veces y agrupadas (CRISPR), y unas proteínas
con actividad endonucleasa (Cas, del inglés “CRISPR-associated sequence”, secuencias asociadas a CRISPR).
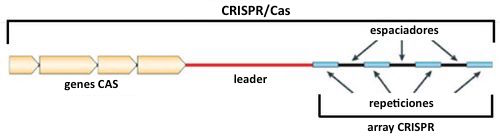
Secuencias CRISPR/Cas en el cromosoma de una bacteria. El sistema incluye un conjunto, denominado array CRISPR, de varias secuencias de DNA espaciadoras alternadas con secuencias palindrómicas (*) repetidas. Estas secuencia repetidas son idénticas y tienen un tamaño entre 21 y 47 pares de bases. Las secuencias espaciadoras tienen un tamaño constante pero su secuencia de DNA es hipervariable y derivan del DNA de bacteriófagos y plásmidos con los que la bacteria ha tenido contacto previamente. Todo el array CRISPR se transcribe como un único RNAm, bajo el control de una secuencia promotora reguladora que se encuentra en la secuencia leader. Además, existen otros genes asociados a CRISPR, denominados genes Cas, del inglés “CRISPR-associated sequences” que codifican para las proteínas Cas con actividad endonucleasa y que cortan el DNA extraño reconocido.
El primer artículo que hace referencia a este sistema es de 1987 (1), en el que analizan el genoma de Escherichia coli y describen por primera vez unas secuencias que se repetían una y otra vez, separadas por otras secuencias a las que denominaron espaciadores. Varios años después, en el 2000, el español Martínez Mójica (2) de la Universidad de Alicante describió esas mismas secuencia en arqueas y las denominó SRSRs, Short Regularly Spaced Repeats. Hoy sabemos que las secuencias CRISPR/Cas se encuentran en aproximadamente el 40% de los genomas de las bacterias secuenciados y el 90% de los de las arqueas. El sistema es más complejo y ya se han descrito de momento tres tipos diferentes de sistemas CRISPR/Cas en procariotas.
Entrevista al investigador Francisco J. Martínez Mójica en El Mundo (29/05/2015)
Mediante el sistema CRISPR/Cas los procariotas son capaces de incorporar
a su genoma un fragmento del DNA invasor que servirá de guía para evitar
futuras invasiones
¿Cómo funciona CRISPR/Cas?
CRISPR/Cas es un sistema de defensa que permite a la bacteria interferir con la infección de un fago. Pero no lo hace ni bloqueando la entrada del fago o de su DNA, ni mediante el sistema enzimático clásico de restricción-modificación, ni mediante un sistema que aborte la infección viral. El sistema es mucho más sofisticado y se activa cuando la bacteria es infectada por un virus que inyecta su DNA extraño al interior de la bacteria. Parte del DNA del virus (o de un plásmido) se incorpora como secuencia espaciador en el genoma de la bacteria entre las secuencias CRISPR.
Posteriormente, se transcribe el array CRISPR y se produce un RNA de interferencia que contiene esa secuencia CRISPR espaciadora incorporada. Este RNA es capaz de guiar a las enzimas Cas hacia una secuencia de DNA complementaria (de un nuevo virus similar al anterior que infecte la bacteria) y degradarlo. Las enzimas Cas, por tanto, son guiadas por el RNA y cortan y empalman el DNA donde le “dice” el RNA.
Es un efectivo sistema de defensa de la bacteria (sistema inmune adaptativo) contra el virus. Podríamos decir que la bacteria queda “vacunada” contra ese virus. Además, la secuencia del virus a destruir queda así almacenada como información en el genoma de la bacteria y de sus descendientes.
CRISPR/Cas es un sistema de defensa bacteriano que escanea el DNA
extraño y si lo reconoce lo destruye
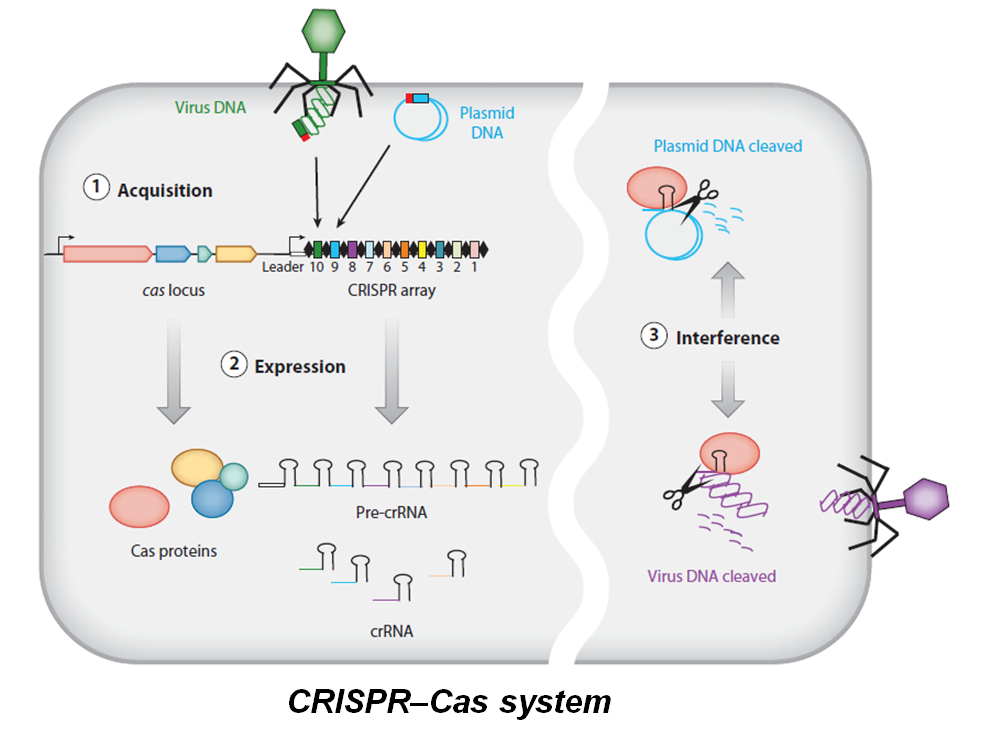
El array CRISPR se transcribe entero como un único RNAm que luego se corta en sitios específicos en cada repetición dando lugar a pequeños RNAs maduros (crRNAs, CRISPR RNA). Cada crRNA contiene una secuencia espaciadora entera más dos pequeños trozos de las regiones repetidas adyacentes. El corte preciso se hace por el complejo de proteínas Cas. Algunas de esta proteínas permanecen unidas al crRNA para formar un agente de defensa activo. La secuencia del espaciador del crRNA es capaz de reconocer y guiar al complejo a la secuencia diana específica, y entonces algunas de la proteínas con actividad endonucleasa cortan el DNA “invasor” . Como todos los espaciadores se transcriben y se procesan en agentes de defensa activos, la bacteria está continuamente en guardia frente a cualquier fago o plásmido invasor.
Una nueva revolución en biología molecular
Después de la invención de la PCR y de los nuevos sistemas de secuenciación masiva, pocos pensaban que habría otra invención que volvería a revolucionar las técnicas de ingeniería genética y biología molecular. Pero el sistema de defensa bacteriano CRISPR/Cas está teniendo unas aplicaciones hasta ahora increíbles. El concepto es simple, modificando la secuencia del crRNA podemos hacer que el sistema corte la secuencia de DNA que nosotros queramos.
En 2012, la estadounidense Jennifer Doudna y la francesa Emmanuelle Charpentier publicaron en la revista Science (3) cómo el sistema CRISPR/Cas puede adaptarse para transformarlo en un método de edición genética muy preciso, eficaz, barato, sencillo, no tóxico, aplicable a todo tipo de DNA, de animales y plantas, que permite modificar el DNA, hacer mutaciones, inserciones, deleciones, y regular la expresión génica para editar y silenciar genomas.
CRISPR/Cas es el mejor “editor de textos” que se ha inventado hasta
ahora para manipular el genoma de cualquier ser vivo
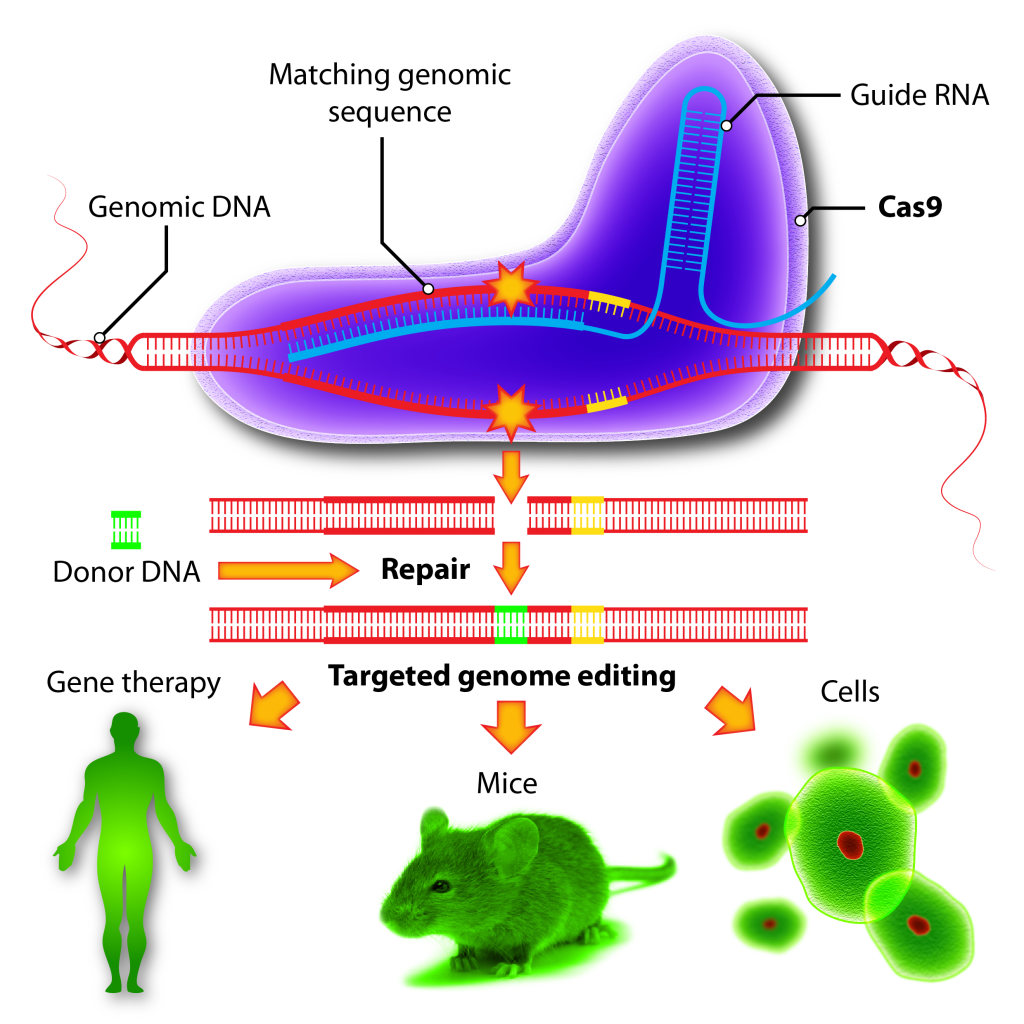
CRISPR/Cas ha transformado ya la tecnología en biología y sus aplicaciones biotecnológicas, biomédicas y de terapia génica son ya una revolución. La posibilidad de manipular la información genética mediante CRISPR/Cas abre la puerta a cambiar radicalmente el tratamiento y la investigación de un gran número de enfermedades y se considera ya uno de los principales hallazgos en la última década de la biomedicina. J. Doudna y E. Charpentier han sido galardonadas con el premio Princesa de Asturias de Investigación Científica y Técnica 2015.
De nuevo la revolución viene de la mano de las bacterias (y arqueas). Primero fueron las enzimas de restricción que actúan como unas “tijeras” capaces de cortar el DNA en sitios específicos. Luego las DNA polimerasas de arqueas permitieron el desarrollo de la amplificación específica de secuencias de DNA mediante la PCR. Y ahora, el sistema de defensa CRISPR/Cas. Ya nadie duda de que el algún día el Nobel premiará estas investigaciones.
Muy recomendable: el sistema CRISPR/Cas explicado por Francis Mojica (48:02)
Video sobre cómo funciona el sistema CASPR/Cas (en inglés, 4:12)
También te puede interesar “La revolución CRISPR/Cas” en Curiosidades de la Microbiología de Manuel Sánchez.
Toda unapágina dedicada a CRISPR del CNB por LLuís Montoliu.
(*) Una secuencia palindrómica es una secuencia de ácido
nucleico (DNA o RNA) que es lo mismo si se lee en una dirección en una hebra
que en la otra dirección en la hebra complementaria, con la que forma una doble
hélice. Por ejemplo:
ATTGGCGGTTAACCGCCAAT
TAACCGCCAATTGGCGGTTA
(1) Nucleotide sequence of the iap gene, responsible foralkaline phosphatase isozyme conversion in Escherichia coli, and identificationof the gene product. Ishino, Y., y col. Journal of Bacteriology, 1987; 169 (12):
5429–5433.
(2) Biological significance of a family of regularly spacedrepeats in the genomes of Archaea, Bacteria and mitocondria. Mojica, F. J., y
col. Molecular microbiology, 2000; 36 (1): 244–246.
(3) A programmable dual-RNA-guided DNA endonuclease in
adaptive bacterial immunity. Jinek, M., y col. Science. 2012; 337 (6096): 816-21.
doi: 10.1126/science.1225829.









Este comentario ha sido eliminado por el autor.
Excelente publicación, falta hablar un poco sobre la secuencia PAM (proto-spacer-associate motif) el cual es una secuencia consenso para reconocimiento por las enzimas CAS. Saludos