El pasado 28 de noviembre de 2025 se confirmó la detección de un brote de peste porcina africana en Cataluña, lo que ha supuesto la primera detección del virus en España desde noviembre de 1994. Como consecuencia, España ha perdido temporalmente el estatus de país libre de la enfermedad que mantenía desde hace treinta años.
Se acaba de publicar el Informe inicial en relación con el brote de peste porcina africana en España, realizado por el Comité Científico Asesor del Ministerio de Agricultura, Pesca y Alimentación.
En dicho informe se analizan las posibles causas de la entrada del virus, su evolución y las medidas a adoptar para su contención. El informe es preliminar y no permite conocer con certeza el origen del brote, pero si documenta la caracterización molecular y sugiere algunas conclusiones que permiten responder a preguntas tan interesantes como que si el virus detectado en noviembre se originó por una liberación accidental desde un centro de investigación.
Caracterización genética del virus
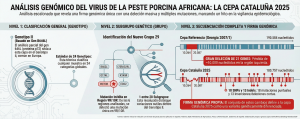
En el informe se describe la caracterización genética del virus detectado en jabalíes en Cataluña en noviembre 2025. Como el genoma de este virus es de gran tamaño (alrededor de 170 genes) y presenta una alta homogeneidad se aplicó el esquema de caracterización de la Unión Europea, que consiste en un análisis escalonado (distintos niveles) que permite aumentar progresivamente la resolución genética para responder a preguntas epidemiológicas complejas.
La primera pregunta es a qué genotipo corresponde el virus (nivel 1). La clasificación global en genotipos se basa en el análisis parcial de un gen concreto, el gen B646L que codifica para la proteína p72. Con esta técnica se han descrito hasta 24 genotipos distintos del virus de la peste porcina africana (VPPA). El virus español fue clasificado dentro del genotipo II, el mismo que lleva años circulando por Europa desde que salió de Georgia en 2007. Esto no sorprendió a los investigadores porque es el único que circula actualmente en Europa. Pero este dato no es suficiente para saber si procede de Italia, Europa del Este o de otro lugar. Es como saber que alguien pertenece a una familia muy grande, pero no saber exactamente de qué rama familiar es.
Análisis genómico del virus de la peste porcina africana: cepa Cataluña 2025. Fuente: Informe inicial en relación con el brote de peste porcina africana en España. Febrero 2025. Ministerio de Agricultura, Pesca y Alimentación. Elaboración propia: Ignacio López-Goñi @microbioblog NotebookLM.
Para afinar más, se realizó un segundo análisis de seis pequeñas regiones genéticas distintas (nivel 2) buscando pequeñas diferencias (mutaciones). Esto permite distinguir hasta 28 subgrupos dentro del genotipo II. El virus español tenía casi el mismo perfil que la cepa de referencia Georgia 2007/1 salvo por una pequeña mutación nunca antes descrita en la región intergénica 9R/10R. Por eso, el aislamiento español corresponde a un nuevo subgrupo genético del genotipo II: el grupo 29.
Para alcanzar la máxima resolución genética, el siguiente paso (nivel 3) supone ya la secuenciación completa del genoma del virus, como si se leyera el libro completo y no solo algunos capítulos. Al compararlo con la cepa de referencia, el genoma del virus español era más corto, presentaba una gran deleción (de unos 10.000 nucleótidos) en la región izquierda del genoma entre las posiciones 10.264–20.087. En otras palabras, el genoma del virus español ha perdido un fragmento grande de su ADN que implica la pérdida de al menos 21 genes. Esos genes no son imprescindibles para que el virus se multiplique, pero sí podrían influir en cómo interactúa con el sistema inmune y en su comportamiento biológico. Además, se identificaron 18 mutaciones puntuales (SNPs, Single Nucleotide Polymorphism) y 13 inserciones y deleciones cortas distribuidas a lo largo de todo el genoma. Esto sugiere que este virus presenta una firma genómica propia y es una variante genéticamente diferenciada del resto de VPPA conocidos hasta ahora.
Comparación con las cepas de laboratorio
Por último, se comparó la secuencia del genoma del virus español con 81 muestras (12 aislados virales históricos de las cepas Georgia 2007 y Armenia 2007 y 69 muestras clínicas procedentes de infecciones experimentales) del laboratorio de investigación CREsA-IRTA, cercano al lugar donde se detectó el brote de la enfermedad en jabalíes en noviembre de 2025.
Los análisis realizados por organismos independientes y mediante metodologías complementarias demostraron que ninguna de las muestras analizadas presentaba la gran deleción de unos 10.000 nucleótidos observada en el virus de campo. Además, tampoco compartían el conjunto de mutaciones distintivas del aislado español. En todas las muestras las secuencias obtenidas eran iguales a las cepas de referencia (Georgia 2007 o Armenia 2007) y no presentaban evidencia de haber adquirido ninguna de las deleciones o mutaciones encontradas en la firma genómica del virus detectado en Cataluña en noviembre de 20025.
Según el informe, estos resultados demuestran que el virus causante del brote de peste porcina africana en Cataluña es genéticamente diferente de los virus empleados en el laboratorio del CReSA-IRTA y por tanto no se originó por una liberación accidental desde el centro de investigación.
No toda Europa está igual de bien vigilada genéticamente
Hasta 2025 se ha notificado la presencia del VPPA en 25 países europeos y solo Bélgica y Suecia han conseguido erradicar el virus de su territorio. Uno de los mayores problemas que existe para los estudios epidemiológicos es que en muchos países del continente europeo no constan datos de la caracterización genética del VPPA, lo que limita mucho la interpretación de su distribución real. Por ejemplo, en países como Hungría, Serbia, Ucrania, Rusia, Armenia, Azerbaiyán, Bielorrusia, Georgia, Lituania o Polonia no hay datos relevantes o son muy limitados de la circulación, dispersión y caracterización el VPPA. Por tanto, el virus aislado en Cataluña en noviembre de 2025 podría ser realmente nuevo… o podría existir en algún país donde no se hayan secuenciado suficientes virus
Entonces, ¿cuál es su origen?
En pocas palabras: no se sabe. Aunque en el informe se discuten varias posibilidades (introducción del virus desde focos activos europeos, la introducción deliberada, o que fuera transportado por actividades humanas), la hipótesis más probable sigue siendo la introducción accidental mediante residuos o restos de alimentos contaminados, la llamada “hipótesis del bocadillo”. El virus es muy resistente en materia orgánica y a bajas temperaturas, y así ha entrado el virus en Europa en varias oleadas en otras ocasiones.
El informe es preliminar y demuestra que es necesario continuar con la vigilancia epidemiológica, la caracterización molecular y la gestión integrada de la fauna silvestre para proteger las explotaciones porcinas y minimizar el impacto sanitario y socioeconómico.