¿Por qué es tan complicado estudiar el genoma de este virus?
Una de las hipótesis sobre el origen del brote de peste porcina africana (PPA) en Cataluña es que el virus proceda del laboratorio del IRTA-CReSA en Bellaterra, ubicado muy cerca de la zona de Collserola donde se han localizado los jabalíes muertos. El origen del brote sería un escape accidental por un fallo de bioseguridad y de custodia, o una liberación intencionada.
Los primeros estudios genómicos han confirmado que el virus de los jabalíes muertos es del genotipo II, el mismo que circula en Europa. Sin embargo, la caracterización molecular por secuenciación de algunos genes sugiere que se trata de una variante (grupo 29) que de momento no parece que esté presente en el medio natural europeo (el virus circulante en Europa parece ser la variante 2-28). La variante 29 es similar a la cepa de referencia que se utiliza con frecuencia en infecciones experimentales en los laboratorios.
(NOTA: hasta donde yo sé, todavía no se han hecho públicos estos análisis y solo se ha emitido una nota de prensa del Ministerio de Agricultura, Pesca y Alimentación del Gobierno de España).
Cabe la posibilidad de que el origen del virus sea por un escape de un laboratorio. Para confirmar estas hipótesis son necesarios análisis comparativos del genoma completo del virus aislado de jabalíes y de las cepas empleadas en el laboratorio para comprobar así si coinciden o no. Seguimos pendientes de estos análisis. Pero, ¿por qué es tan complicado estudiar el genoma de este virus?
1. Un genoma grande y complejo
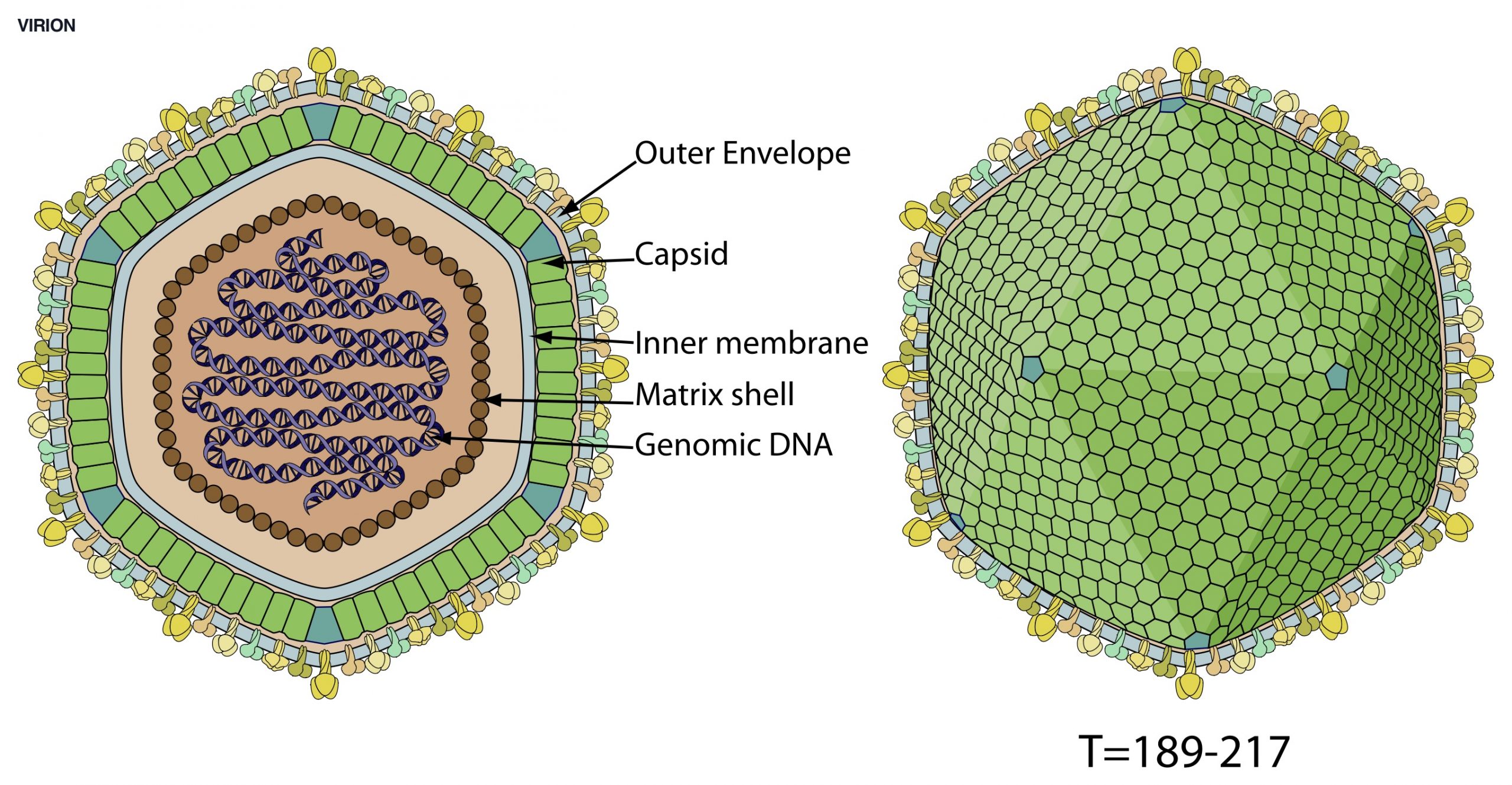
El genoma del virus PPA es una molécula lineal de ADN de doble hebra de entre 170-190 mil pares de bases, con regiones repetidas en sus extremos altamente variables. Es un genoma complejo y difícil de secuenciar. El virus tiene más de 180 proteínas, muchas de las cuales todavía no sabe qué función tienen. La longitud del genoma y el número de genes pueden variar según la cepa del virus. El virus de la gripe tiene solo 8 segmentos genómicos de ARN de unos 14 mil pares de bases totales y poco más de una docena de proteínas.
2. Hay pocas secuencias en bases de datos
En las bases de datos solo se han depositado 348 genomas completos o casi completos. Esto limita mucho la capacidad de comparar cepas y de comprender la evolución y distribución del virus. Del virus de la gripe hay más de 129.000 genomas secuenciados.
3. El genotipado se hace de solo unos genes concretos
Secuenciar el genoma completo requiere mucho tiempo, mano de obra y es costoso. Por eso, la epidemiología molecular se hace por comparación de secuencias de genes concretos, de pequeños fragmentos que representan menos del 1% del genoma del virus. Actualmente, los virus PPA se clasifican en 24 genotipos según la región terminal variable del gen B646L, que codifica la proteína estructural p72 , la proteína principal de la cápside, cubierta exterior del virus.
4. Es un virus peligroso de manipular
El virus PPA no es patógeno para el ser humano, pero es muy contagioso y roza el 100% de mortalidad en cerdos. Puede tener consecuencias devastadoras en el ganado porcino y el perjuicio económico que puede ser tremendo. Por eso, la manipulación del virus requiere laboratorios de alta seguridad (BSL-3), y solo unos pocos centros están equipados para manipular muestras y aislar el virus.
Analizar el genoma del virus PPA es complicado, pero no imposible. Es necesario aislar el virus de muestras de los jabalíes, secuenciar el genoma completo y compararlo con el de las cepas empleadas en el laboratorio. La comunicación debe transmitir confianza y para eso debe ser transparente, clara, sencilla y rigurosa. Necesitamos ese análisis cuanto antes.
NOTA (2/1/2026): Aunque todavía no hay datos públicos y a falta de la confirmación definitiva, el Gobierno catalán ha informado de los resultados preliminares de la secuenciación del virus aislado de los jabalíes y de su comparación con 19 muestras de virus del laboratorio. Estos análisis demuestran que el virus de los jabalíes presenta hasta 27 mutaciones y una pérdida (deleción) de parte del genoma, diferentes de los virus empleados en el laboratorio del CReSA-IRTA y de las secuencias del virus de distintos países existentes en las bases de datos. Esto sugiere que se trata de una cepa del virus nueva no descrita hasta ahora, quizá menos virulenta. Según esta información, el virus que ha causado el brote en Cataluña no salió del laboratorio y refuerza de nuevo la «hipótesis del bocadillo«: que el origen del virus fuera por un alimento o material contaminado probablemente con una cepa no detectada hasta ahora, quizá de algún país europeo donde existe propagación del virus, no haya un control epidemiológico estricto y esta cepa concreta haya podido pasar desapercibida.